Chapter 4 Datenkunde und Datenaufbereitung
In diesem Kapitel geht es um den auf den ersten Blick unspannendsten Teil der Forschung: Datenaufbereitung und -management. Gleichzeitig ist es einer der wichtigsten Schritte: ohne Daten können viele Forschungsfragen nicht angemessen beantwortet werden. Abbildung 4.1 zeigt den typischen Arbeitsablauf eines Forschungsprojekts.
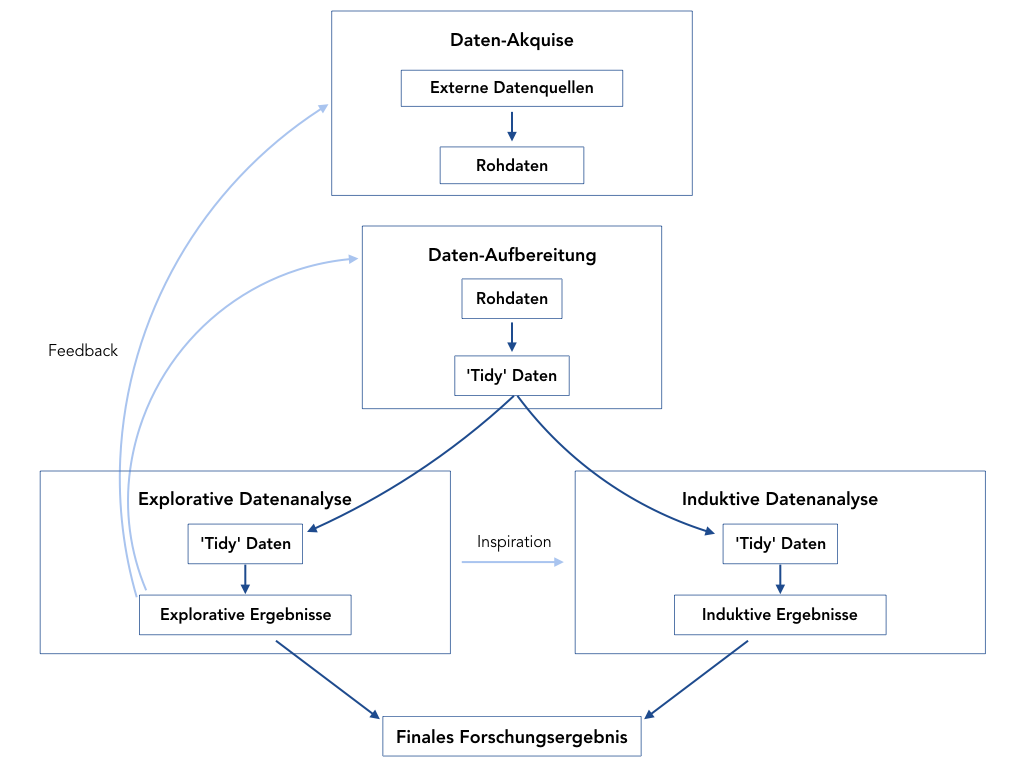
Abbildung 4.1: Typischer Arbeitsablauf eines Forschungsprojekts
In diesem Kapitel liegt der Fokus auf den ersten beiden Abschnitten, der Akquise und der Aufbereitung Ihrer Daten. Laut dieser Umfrage verwenden Datenspezialist*innen regelmäßig 80% ihrer Arbeitszeit auf diese beiden Schritte. Um hier also Zeit und Nerven zu sparen ist es wichtig, sich mit den grundlegenden Arbeitsschritten und Algorithmen vertraut zu machen. Zum Glück ist R sehr gut zur reproduzierbaren Datenaufbereitung geeignet und stellt dank vieler hilfreicher Pakete eine große Hilfe in diesem wichtigen Prozess dar.
Ein zentrales Anliegen dieses Abschnitts liegt darin, Ihnen Methoden zur reproduzierbaren und transparenten Datenaufbereitung an die Hand zu geben. Für eine glaubwürdige Forschungsarbeit ist es unerlässlich, dass der Weg von der Datenerhebung hin zum Forschungsergebnis, also der gesamte Prozess in der obigen Abbildung, transparent und nachvollziehbar ist. Daher muss der Datenaufbereitungsprozess gut dokumentiert werden. Dank skriptbasierter Sprachen wie R ist das im Prinzip ein Kinderspiel.
Wenn Sie nämlich alle Arbeitsschritte nach der Datenerhebung in R durchführen, müssen Sie einfach nur Ihre Skripte aufheben - und schon haben Sie die beste Dokumentation, die man sich wünschen kann. Das Wichtigste bei diesem Prozess: Sie dürfen nie die Rohdaten selbst verändern.
Alle Änderungen an den Rohdaten müssen durch ein R Skript vorgenommen werden, und die veränderten Daten müssen unter neuem Namen gespeichert werden. Wenn Sie sich das einmal angewöhnt haben, können Sie nicht nur vollkommen transparent in Ihrer Forschung sein, Sie können auch nicht aus Versehen und unwiderruflich Ihre wertvollen Rohdaten zerstören.
Und wenn Sie sich mit den grundlegenden Algorithmen einmal vertraut gemacht haben kann Datenaufbereitung wider Erwarten auch wirklich Spaß machen!
Dieses Kapitel folgt dem typischen Arbeitsablauf eines Forschungsprojektes und beschäftigt sich mit den ersten beiden Abschnitten aus der obigen Grafik, der Daten-Akquise und der Daten-Aufbereitung, wobei Letztere im Mittelpunkt stehen soll. Entsprechend ist das Kapitel folgendermaßen strukturiert:
Als erstes werden wir uns einen Überblick über die verschiedenen Arten von Daten verschaffen. Danach geht es los mit der Datenakquise. Hier lernen wir, wie man Daten aus häufig verwendeten Datenbanken direkt über R herunterlädt. Als nächstes werden Funktionen zum Lesen und Schreiben von Datensätzen und typische Herausforderungen in diesem Prozess besprochen. Danach kommt ein sehr umfangreicher Block zum Thema Datenaufbereitung, in dem Sie lernen, wie Sie Ihre Rohdaten in ein Format überführen, das für die statistische Analyse geeignet ist. Zum Abschluss des Kapitels wird noch die Rolle des Datenmanagements für transparente Forschung verdeutlicht und auf die Debatten über die Ko-Existenz verschiedener Pakete für die Datenaufbereitung in R hingewiesen.
Verwendete Pakete
library(countrycode)
library(here)
library(WDI)
library(tidyverse)
library(data.table)
library(R.utils)
library(haven)Hinweis: In diesem Kapitel verwenden wir für die Arbeit mit Daten vor allem Pakete aus dem sogenannten tidyverse. Ich habe mich für diese Pakete entschieden, weil sie meiner Meinung nach die für R-Beginner am einfachsten zu lernenden Pakete sind und sie zu sehr einfach zu lesendem Code führen. Zudem sind sie sehr weit verbreitet. Es gibt aber auch sehr gute Alternativen und gerade für sehr große Datensätze kommen Sie nicht an dem Paket data.table vorbei. Die Rolle des
tidyverseund der Debatte um die Pakete in R wird am Ende des Kapitels beschrieben. Bis dahin verweise ich häufig auf weitere Quellen, in denen die Implementierung der Arbeitsschritte in anderen Paketen als demtidyversebeschrieben wird.
4.1 Arten von Daten
Es gibt verschiedene mehr oder weniger konsistente Klassifizierungen von Daten, die jeweils auf unterschiedliche Aspekte von Daten oder auch Variablen abzielen.
Eine sehr prominente Unterscheidung wird zwischen quantitativen und qualitativen Daten getroffen. Bei quantitativen Daten handelt es sich grob gesagt um numerische Daten, also Daten, die Sie in Zahlen ausdrücken können. ‘Größe’, ‘Preis’, ‘BIP’ oder ‘Gehalt’ sind typische Beispiele. Qualitative Daten werden intuitiv nicht-numerisch ausgedrückt. Häufig handelt es sich um text-basierte oder beschreibende Daten. In der Praxis werden Sie aber merken, dass die Grenze zwischen quantitativen und qualitativen Daten häufig deutlich schwammiger ist, als man das auf den ersten Blick glauben möchte, denn häufig werden qualitative Beschreibungen quantifiziert und dann mit typischen quantitativen Methoden analysiert. Auch werden sogenannte mixed methods-Ansätze immer beliebter, welche qualitative und quantitative Methoden kombinieren.
Vor allem in der Psychologie unterscheidet man zwischen manifesten und latenten Variablen. Manifeste Variablen sind direkt beobachtbar und ihre Bedeutung ist häufig klar. Die Körpergröße ist z.B. eindeutig messbar und jede*r weiß was damit gemeint ist.
Latente Variablen sind nicht direkt beobachtbar und sind häufig erklärungsbedürftig. Nutzen ist zum Beispiel nicht beobachtbar.14 Zudem muss in der Regel erst einmal deutlich gemacht werden, was mit dem Begriff genau gemeint ist.
Ein großer Teil von Forschungsarbeit ist die Operationalisierung einer latenten Variable durch eine oder mehrere manifeste Variablen. Wir sprechen dann davon, dass eine oder mehrere manifeste Variablen als Indikator für eine latente Variable verwendet werden. Wirtschaftliche Entwicklung z.B. ist als solche nicht direkt beobachtbar und wird häufig durch das BIP operationalisiert.15
Der Human Development Index ist der Versuch, wirtschaftliche Entwicklung durch mehr als eine manifeste Variable zu operationalisieren, also durch beobachtbare Variablen messbar zu machen. Eine solche Operationalisierung ist natürlich immer kritisch zu hinterfragen und ist nicht selten ein Einfallstor für subjektive und manchmal auch manipulative Wertentscheidungen.
In der Praxis sehr relevant ist zudem die Unterscheidung der vier Skalenniveaus von Daten, da die Art der Skala bestimmt, welche Methode angemessen ist um die Daten zu analysieren. Hier wird zwischen nominal, ordinal, intervall und verhältnis skalierten Daten unterschieden, wobei intervall- und verhältnisskalierte Daten häufig unter dem Label kardinalskalierte Daten zusammen gefasst werden, wie aus der Abbildung 4.2 hervorgeht:
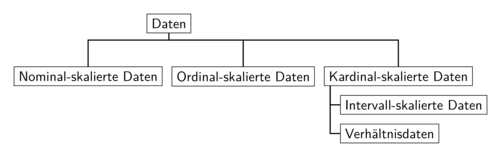
Abbildung 4.2: Skalenniveaus von Daten
Wir sprechen von nominalskalierten Daten wenn wir den einzelnen Ausprägungen der Daten zwar bestimmte Werte oder eindeutige Beschreibungen zuordnen können, diese aber keine natürliche Rangfolge aufweisen. So können wir einer Person eine Haarfarbe zuordnen, allerdings die verschiedenen Haarfarben in keine natürliche Rangfolge einordnen. Ebenso können wir z.B. Tiere in die Kategorien “Hund”, “Katze” und “Sonstiges” einordnen, aber eine natürliche Rangfolge dieser Kategorien gibt es nicht. Als Konsequenz können wir die einzelnen Ausprägungen zwar zählen, aber sonst keine komplexeren mathematischen Operationen - wie z.B. die Berechnung eines Mittelwerts - ausführen.
In R werden solche Daten in der Regel als character oder als factor beschrieben.
In der Regel ist es einfacher die Daten erst als character zu erstellen und
dann mit der Funktion factor() in Faktoren umzuwandeln:
beobachtete_haarfarben <- c("Blond", "Braun", "Schwarz",
"Blond", "Braun", "Braun")
typeof(beobachtete_haarfarben)#> [1] "character"beobachtete_haarfarben <- factor(beobachtete_haarfarben)
beobachtete_haarfarben#> [1] Blond Braun Schwarz Blond Braun Braun
#> Levels: Blond Braun SchwarzBeachten Sie, dass Faktoren besondere Arten von integer sind.
Um zu testen, ob eine Variable als Faktor kodiert ist, können Sie die Funktion
is.factor() verwenden:
typeof(beobachtete_haarfarben)#> [1] "integer"is.factor(beobachtete_haarfarben)#> [1] TRUEDie einzelnen Ausprägungen eines Faktors können mit der Funktion table gezählt
werden. Der häufigste wird dabei ‘Modus’ genannt:
table(beobachtete_haarfarben)#> beobachtete_haarfarben
#> Blond Braun Schwarz
#> 2 3 1Mehr Informationen zur Arbeit mit Objekten des Typs factor finden Sie
in Abschnitt 3.3.9.
Bei ordinalskalierten Daten können die einzelnen Ausprägungen in eine klare Rangfolge gebracht werden, aber die Abstände sind nicht sinnvoll interpretierbar. Das klassische Beispiel sind Schulnoten: eine ‘1’ ist besser als eine ‘2’, aber weder ist eine 1 ‘doppelt so gut’ wie eine 2, noch sind zwei einser genauso gut wie eine 2.
Ordinalskalierte Daten werden in R am besten auch als factor behandelt, allerdings
müssen Sie die Reihenfolge explizit spezifizieren indem Sie die Level über das
Argument levels explizit angeben und dem Argument ordered den Wert TRUE
übergeben:
noten <- c(rep(1, 3), rep(2, 4), rep(3, 6), rep(4, 2), rep(5, 3))
noten#> [1] 1 1 1 2 2 2 2 3 3 3 3 3 3 4 4 5 5 5noten <- factor(noten, levels = 1:6, ordered = T)
noten#> [1] 1 1 1 2 2 2 2 3 3 3 3 3 3 4 4 5 5 5
#> Levels: 1 < 2 < 3 < 4 < 5 < 6Dass der Faktor geordnet ist erkennen wir daran, dass bei der Auflistung der
Levels das Symbol < verwendet wird um die Reihenfolge zu illustrieren.
Um bei bestehenden Faktoren die Reihenfolge zu spezifizieren, verwenden Sie die
Funktion ordered() (das und weitere technische Besonderheiten von Objekten des
Typs factor werden in Abschnitt 3.3.9 genauer beschrieben):
noten <- factor(noten, levels = 1:6, ordered = F)
noten#> [1] 1 1 1 2 2 2 2 3 3 3 3 3 3 4 4 5 5 5
#> Levels: 1 2 3 4 5 6noten <- ordered(noten, levels = 1:6)
noten#> [1] 1 1 1 2 2 2 2 3 3 3 3 3 3 4 4 5 5 5
#> Levels: 1 < 2 < 3 < 4 < 5 < 6Da wir ordinal-skalierte Daten ordnen können, ist es hier z.B. auch möglich mit
Hilfe der Funktion quantile() empirische Quantile zu berechnen:16
quantile(noten, type = 1)#> 0% 25% 50% 75% 100%
#> 1 2 3 4 5
#> Levels: 1 < 2 < 3 < 4 < 5 < 6Bei intervallskalierten Daten können wir die Ausprägungen nicht nur in eine
Rangfolge bringen, sondern auch die Abstände zwischen den Ausprägungen sinnvoll
interpretieren.
Während es bei Noten also keinen Sinn ergibt, mathematische Operationen wie ‘Addition’
oder ‘Substraktion’ zu verwenden (und die Abstände entsprechend nicht konsistent zu
interpretieren sind), ist dies bei intervallskalierten Daten wie z.B. Jahreszahlen
möglich:
zwischen 1999 und 2005 liegt der gleiche Abstand wie zwischen 2009 und 2015.
Entsprechend werden intervallskalierte Daten in in der Regel als integer oder
double gespeichert und wir können Kennzahlen wie den Mittelwert oder die Varianz
berrechnen.
Allerdings verfügen intervallskalierte Daten über keinen absoluten Nullpunkt, sodass Divisionen und Multiplikationen keinen Sinn machen. Das ist bei verhältnisskalierten Daten wie Gewicht, Preis oder Alter anders. Das kann man am besten an folgendem Beispiel illustrieren:
Beispiel: Inverall- vs. verhältnisskalierte Temperaturen Wenn wir die Temperatur in Grad Celsius messen haben wir eine Skala ohne absoluten Nullpunkt. Entsprechend können wir nicht sagen, dass 40 Grad Celsius doppelt so warm sind wie 20 Grad Celsius, nur das der Abstand der Gleiche ist wie zwischen 10 und 30 Grad Celsius. Das wird deutlich, wenn wir uns fragen ob 10 Grad Celsius doppelt so warm wären wie -10 Grad Celsius. Eine Lösung ist die Temperatur in Kelvin anzugeben, denn für Kelvin ist ein absoluter Nullpunkt definiert. Entsprechend können wir auch sagen, dass 20 Kelvin halb so warm ist wie 40 Kelvin - wobei beides ziemlich kalt wäre.
Da sowohl intervall- als auch verhältnisskalierte Daten als double oder integer
repräsentiert werden, ist Vorsicht geboten: wir müssen immer selbst entscheiden
welche Maße wir für die Daten berechnen und R gibt uns keinen Fehler aus, wenn
wir für zwei intervallskalierte Variablen ein Verhältnis berechnen wollen.
Tabelle 4.1 gibt nochmal einen Überblick über die Skalenniveaus und die dazugehörigen Objekte. Wie oben erwähnt bestimmt das Skalenniveau die anwendbaren statistischen Operationen und Maße. Tabelle 4.2 fasst das für die uns bislang bekannten statistischen Maße kurz zusammen. Alle diese Konzepte werden im Kapitel 8 vertiefter behandelt.
| Skalenniveau | Beispiel | Messbare Eigenschaften | Typisches R Objekt |
|---|---|---|---|
| Nominal | Haarfarbe, Telefonnummer | Häufigkeit | character, factor |
| Ordinal | Schulnote, Zufriedenheit | Häufigkeit, Rangfolge | factor |
| Intervall | Temperatur in C\(^\circ\), Jahreszahl | Häufigkeit, Rangfolge, Abstand | integer, double |
| Verhältnis | Preise, Alter | Häufigkeit, Rangfolge, Abstand, abs. Nullpunkt | integer, double |
| Nominal | Ordinal | Intervall | Verhältnis | |
|---|---|---|---|---|
| Modus | \(\checkmark\) | \(\checkmark\) | \(\checkmark\) | \(\checkmark\) |
| Quantile | \(\times\) | \(\checkmark\) | \(\checkmark\) | \(\checkmark\) |
| Interquantilsabstand | \(\times\) | \(\checkmark\) | \(\checkmark\) | \(\checkmark\) |
| Rankkorrelation | \(\times\) | \(\checkmark\) | \(\checkmark\) | \(\checkmark\) |
| Mittelwert | \(\times\) | \(\times\) | \(\checkmark\) | \(\checkmark\) |
| Varianz | \(\times\) | \(\times\) | \(\checkmark\) | \(\checkmark\) |
| Pearson-Korrelation | \(\times\) | \(\times\) | \(\checkmark\) | \(\checkmark\) |
Wahrscheinlich kennen Sie auch noch die Unterscheidung zwischen diskreten und stetigen Werten. Diese Kategorisierungen ist nicht vollkommen konsistent mit den Skalenniveaus: zwar sind kardinale Daten in der Tendenz eher stetig und nominale, bzw. ordinale Daten eher diskret, allerdings gibt es auch diskrete kardinale Daten (aber keine stetigen nominalen Daten).
Hinweis zum Angeben: Aus der Skalierung oben wird ersichtlich, dass man mit ordinalskalierten Daten keine Durchschnitte bilden darf - man kann sie ja noch nicht einmal addieren. Ein Bereich wo dieser fundamentalen Regel ständig Gewalt angetan wird ist die Schule: wer hat noch nie von einer Durchschnittsnote gehört? Zum Glück gehört das an der Universität der Vergangenheit an…
4.2 Datenakquise
Der erste Schritt in der Arbeit mit Daten ist immer die Akquise der Daten. Je nach verwendeter Methode und Fragestellung ist das mehr oder weniger Arbeit. Im einfachsten Fall sind die von Ihnen benötigten Daten bereits erhoben und über das Internet frei zugänglich. Das trifft z.B. auf viele makroökonomische Indikatoren, wie das BIP, den Gini-Index oder die Arbeitslosigkeit zu. In diesem Falle müssen Sie einfach nur noch die passende Quelle finden,17 laden die Daten herunter und machen beim nächsten Schritt zum Einlesen von Datensätzen weiter, oder überlegen ob sie die Daten sogar direkt mit R herunterladen wollen.
4.2.1 Exkurs 1: Ländercodes übersetzen
Gerade wenn Sie mit makroökonomischen Daten arbeiten werden Sie häufig in Kontakt
mit Ländercodes kommen.
In vielen Datensätzen werden Länder unterschiedlich abgekürzt.
So mögen manche Datensätze zwar ausgeschriebene Ländernamen wie “Deutschland”
verwenden, andere verwenden aber eher den
iso3c-Code
“DEU”, während wieder andere den
iso2c-Code
“DE” verwenden.
Wenn Sie sich dann Daten vom IWF herunterladen wundern Sie sich vielleicht, dass
Deutschland dort mit der Zahl 134 kodiert wird.
Zum Glück gibt es ein R-Paket, das die Übersetzung der Codes kinderleicht macht:
countrycode (Arel-Bundock, Enevoldsen, und Yetman 2018).
Es stellt Ihnen unter anderem die Funktion countrycode() zur Verfügung, mit der
Sie die Codes einfach übersetzen können. Die Funktion benötigt die folgenden
Argumente:
sourcevar akzeptiert einen character oder einen Vektor mit den zu
übersetzenden Ländercodes.
origin gibt die Form dieser Codes an und destination spezifiziert den Code
in den Sie die sourcevar übersetzen wollen.
Die Abkürzungen finden Sie in der Hilfefunkion von countrycode().
Nehmen wir einmal an, wir möchten die iso2c-Codes für Frankreich und die
Schweiz herausfinden. Das geht folgendermaßen:
countrycode::countrycode(
sourcevar = c("Frankreich", "Schweiz"),
origin = "country.name.de",
destination = "iso3c")#> [1] "FRA" "CHE"In diesem Fall verdeutlicht origin="country.name.de", dass wir die Originalnamen
auf Deutsch angegeben haben und destination="iso3c" dass wir in iso3c
übersetzen wollen.
Wenn wir wissen wollen welches Land sich hinter der IWF Nummer 112 verbirgt
schreiben wir:
countrycode::countrycode(
sourcevar = c("112"),
origin = "imf",
destination = "country.name.de")#> [1] "Großbritannien"Die Funktion countrycode() kennt bereits alle wichtigen Ländercodes.
Schauen Sie in der Hilfefunktion nach wie die Codes abgekürzt werden.
Grundsätzlich empfehle ich Ihnen in Ihrer Arbeit möglichst auf das Ausschreiben
von Ländernamen zu verzichten und stattdessen mit eindeutigen Kürzeln zu
arbeiten.
Ich arbeite z.B. immer mit den iso3c-Codes, da sie trotz Abkürzung sehr intuitiv
lesbar sind.
Das Problem mit ausgeschriebenen Ländernamen lässt sich anhand der
Tschechischen Republik gut verdeutlichen.
Der iso3c-Code ist hier eindeutig CZE, allerding verwenden manche Datenbanken
den Namen ‘Czechia’ und andere ‘Czech Republik’.
Das countrycode-Paket übersetzt beide Namen in CZE:
countrycode::countrycode("Czech Republic", "country.name", "iso3c")#> [1] "CZE"countrycode::countrycode("Czechia", "country.name", "iso3c")#> [1] "CZE"Das kann manchmal zu Problemen beim Zusammenführen von Datensätzen führen, da R nicht von sich aus weiß, dass ‘Czechia’ und ‘Czech Republik’ das gleiche Land bezeichnen. Da die Ländercodes immer eindeutig sind empfehle ich daher immer mit den Kürzeln zu arbeiten und beim ersten Übersetzen immer besonders vorsichtig zu sein.
Das Anwendungsgebiet von countrycode() geht übrigens weit über das Übersetzen
von Länderkürzeln hinaus: manchmal möchten Sie vielleicht eine ganz andere
Übersetzung durchführen. In einem solchen Falle können Sie
countrycode() über das Argument custom_dict auch einen data.frame mit
dem neuen Code übergeben und die Funktion ansonsten äquivalent nutzen.
Das kann z.B. passieren wenn Sie Daten von einer Quelle verwenden, die Länder
oder andere Beobachtungen nach einer eigenen Klassifizierung kodiert und diese
Kodierung durch eine eigene Tabelle beschreibt.
Im folgenden Beispiel ist genau das der Fall: wie haben Daten über die Exporte
verschiedener Güterarten. Die Güter sind jedoch nach dem Harmonized System
der UN kodiert (siehe
hier):
head(export_data)#> Good Exports
#> 1: 1 43592
#> 2: 3 234293
#> 3: 5 23842
#> 4: 6 123103Wir können uns aber eine Korrespondenztabelle herunterladen, welche die Codes in konkrete Beschreibungen übersetzt. Eine solche Tabelle sieht folgendermaßen aus:
head(correspondence_table, 3)#> Code Description
#> 1: 1 Food and beverages
#> 2: 2 Industrial supplies nes
#> 3: 3 Fuels and lubricantsWir können nun die Funktion countrycode() zur Übersetzung der Codes in ihre
Beschreibung verwenden (dabei müssen Tabellen in anderen Formaten immer mit
as.data.frame() in einen Data Frame umgewandelt werden):
export_data <- dplyr::mutate(
export_data,
Good_Description=countrycode(
Good, "Code", "Description",
custom_dict = as.data.frame(correspondence_table)
)
)
export_data#> Good Exports Good_Description
#> 1: 1 43592 Food and beverages
#> 2: 3 234293 Fuels and lubricants
#> 3: 5 23842 Transport equipment, and parts and accessories thereof
#> 4: 6 123103 Consumption goods4.2.2 Exkurs 2: Daten direkt mit R herunterladen
Manchmal können Sie sich viel Arbeit sparen indem Sie die Daten direkt in R über eine so genannte API herunterladen. Das bedeutet, dass Sie über R einen direkten Zugang zum Server mit den Daten herstellen und die Daten direkt in R einladen. Das hat den Vorteil, dass die Daten in der Regel bereits in einem gut weiterzuverarbeitenden Zustand sind und dass aus Ihrem Code unmittelbar ersichtlich wird wo Ihre Rohdaten herkommen.18
Es lohnt sich daher, gerade wenn Sie aus einer Quelle mehrere Daten beziehen wollen, nachzuschauen ob ein R Paket oder eine besondere API verfügbar ist. Im Folgenden möchte ich das Vorgehen mit dem Paket WDI (Arel-Bundock 2019) illustrieren, welches Ihnen Zugriff auf die Weltbankdaten ermöglicht.
Das Paket WDI stellt Funktionen sowohl zum Suchen als auch zum direkten
Download von Daten aus der Datenbank der Weltbank zur Verfügung.
Diese Datenbank ist extrem nützlich, weil sie makroökonomische Indikatoren
für die ganze Welt aus verschiedenen Quellen bündelt.
Als erstes müssen Sie den Code des von Ihnen gewünschten Indikators herausfinden. Dazu gehen Sie am besten auf die Startseite der Weltbankdatenbank und suchen dort nach den Indikatoren ihrer Wahl. Nehmen wir einmal an, Sie wollen Daten zum Export und zur Arbeitslosigkeit für Deutschland und Österreich für die Jahre 2012-2014 haben.
Sie suchen also nach den Indikatoren und lesen den Code aus der URL des Indikators ab, wie in Abbildung 4.3 dargestellt:19
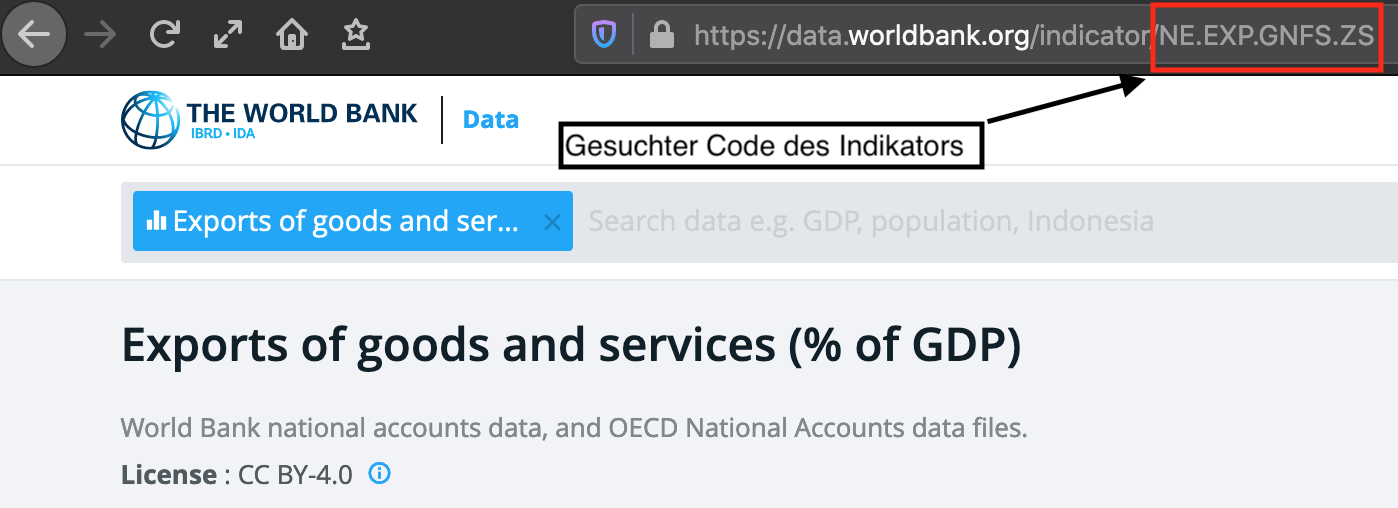
Abbildung 4.3: Ablesen des Codes aus der URL
Über die Weltbankseite finden Sie heraus, dass die beiden von Ihnen gesuchten
Indikatoren mit NE.EXP.GNFS.ZS und SL.UEM.TOTL.ZS kodiert sind.
Nun verwenden Sie die Funktion WDI::WDI() um direkt auf die Daten zuzugreifen.
Die Funktion benötigt dabei die folgenden Argumente:
country verlangt nach einem Vektor mit Länderkürzeln.
Der countrycode-Code für die von der Weltbank geforderten Kürzel ist wb und
es ist entsprechend einfach diesen Vektor zu erstellen.
Das zweite relevante Argument ist indicator und benötigt einen Vektor der
gewünschten Indikatoren.
Über die Argumente start und stop geben Sie das erste und letzte gewünschte
Beobachtungsjahr an.
Die weiteren Argumente sind nicht von unmittelbarem Interesse.
Nun können Sie die Funktion WDI::WDI() folgendermaßen verwenden um Export-
und Arbeitslosendaten für Deutschland und Österreich zwischen 2012 und 2014 zu
bekommen:
t_beginn <- 2012
t_ende <- 2014
laender <- countrycode::countrycode(c("Germany", "Austria"),
"country.name", "wb")
indikatoren <- c("NE.EXP.GNFS.ZS", "SL.UEM.TOTL.ZS")
daten <- WDI::WDI(
country = laender,
indicator = indikatoren
start = t_beginn,
end = t_ende
)daten#> iso2c country year NE.EXP.GNFS.ZS SL.UEM.TOTL.ZS
#> 1: AT Austria 2012 53.97368 4.865
#> 2: AT Austria 2013 53.44129 5.335
#> 3: AT Austria 2014 53.38658 5.620
#> 4: DE Germany 2012 45.98254 5.379
#> 5: DE Germany 2013 45.39788 5.231
#> 6: DE Germany 2014 45.64482 4.981Mit derlei Paketen können Sie sich häufig viel Zeit sparen, insbesondere wenn Sie mehrere Datensätze von der gleichen Quelle benötigen.
4.3 Daten einlesen und schreiben
4.3.1 Einlesen von Datensätzen
Wenige Arbeitsschritte können so frustrierend sein wie das Einlesen von Daten. Sie können sich gar nicht vorstellen was hier alles schiefgehen kann! Aber kein Grund zur übertriebenen Sorge: wir können viel Frustration vermeiden wenn wir am Anfang unserer Karriere ausreichend Zeit in die absoluten Grundlagen von Einlesefunktionen investieren. Also, auch wenn die nächsten Zeilen etwas trocken wirken: sie werden Ihnen später viel Zeit ersparen!
Das am weitesten verbreitete Dateiformat ist csv. ‘csv’ steht für ‘comma separated values’ und diese Dateien sind einfache Textdateien, in denen Spalten mit bestimmten Symbolen, in der Regel einem Komma, getrennt sind. Aufgrund dieser Einfachheit sind diese Dateien auf allen Plattformen und quasi von allen Programmen ohne Probleme lesbar.
In R gibt es verschiedene Möglichkeiten csv-Dateien einzulesen.
Die mit Abstand beste Option ist dabei die Funktion data.table::fread() aus dem Paket
data.table, da sie nicht nur sehr flexibel spezifiziert werden kann, sondern
auch deutlich schneller als andere Funktionen arbeitet.
Wir gehen im Folgenden davon aus, dass wir die Datei data/tidy/export_daten.csv
einlesen wollen.
Die Datei sieht folgendermaßen aus:
iso2c,year,Exporte
AT,2012,53.97
AT,2013,53.44
AT,2014,53.38Es handelt sich also um eine sehr standardmäßige csv-Datei, die wir einfach
mit der Funktion data.table::fread() einlesen können.
Dazu übergeben wir data.table::fread() nur das einzige wirklich notwendige Argument:
den Dateipfad.
Der besseren Übersicht halber sollte dieser immer separat definiert werden:
daten_pfad <- here::here("data/tidy/export_daten.csv")
daten <- data.table::fread(daten_pfad)
daten#> iso2c year Exporte
#> 1: AT 2012 53.97
#> 2: AT 2013 53.44
#> 3: AT 2014 53.38Vielleicht fragen Sie sich wie data.table::fread() die Spalten bezüglich ihres
Datentyps interpretiert hat?
Das können wir folgendermaßen überprüfen:
typeof(daten[["year"]])#> [1] "integer"In der Regel funktioniert die automatische Typerkennung von data.table::fread() sehr gut.
Ich empfehle dennoch die Typen im Zweifel manuell zu spezifizieren, aus folgenden
Gründen: (1) Sie merken leichter wenn es mit einer Spalte ein Problem gibt,
z.B. wenn in einer Spalte, die ausschließlich aus Zahlen besteht ein Wort
vorkommt. Wenn Sie diese Spalte nicht manuell als double spezifizieren würden,
würde data.table::fread() sie einfach still und heimlich als character interpretieren und Sie
wundern sich später, warum Sie für die Spalte keinen Durchschnitt berechnen können;
(2) Ihr Code wird leichter lesbar; und (3) der Einlesevorgang wird deutlich
beschleunigt da data.table::fread() die Typen nicht selbst ‘erraten’ muss.
Sie können die Spaltentypen manuell über das Argument colClasses einstellen,
indem Sie einfach einen Vektor mit den Datentypen angeben:
daten_pfad <- here::here("data/tidy/export_daten.csv")
daten <- data.table::fread(
daten_pfad, colClasses = c("character", "double", "double")
)
typeof(daten[["year"]])#> [1] "double"Da es bei sehr großen Dateien einen extremen Unterschied macht ob Sie die
Spaltentypen angeben oder nicht macht es in einem solchen Fall häufig Sinn,
zunächst mal nur die erste Zeile des Datensatzes einzulesen, sich
anzuschauen welche Typen die Spalten haben sollten und dann den gesamten
Datensatz mit den richtig spezifizierten Spaltentypen einzuladen.
Sie können nur die erste Zeile einladen indem Sie das Argument nrows
verwenden:
daten_pfad <- here("data/tidy/export_daten.csv")
daten <- data.table::fread(
daten_pfad, colClasses = c("character", "double", "double"),
nrows = 1)
daten#> iso2c year Exporte
#> 1: AT 2012 53.97Manchmal möchten Sie auch nur eine bestimmte Auswahl an Spalten einlesen.
Auch das kann bei großen Datensätzen viel Zeit sparen.
Wenn wir nur das Land und die Anzahl der Exporte haben wollen, spezifizieren wir
das über das Argument select:
daten_pfad <- here::here("data/tidy/export_daten.csv")
daten <- data.table::fread(
daten_pfad, colClasses = c("character", "double", "double"),
nrows = 1,
select = c("iso2c", "Exporte")
)
daten#> iso2c Exporte
#> 1: AT 53.97Die Beispiel-Datei oben war sehr angenehm formatiert. Häufig werden aber andere
Spalten- und Dezimalkennzeichen verwendet.
Gerade in Deutschland ist es verbreitet, Spalten mit ; zu trennen und das
Komma als Dezimaltrenner zu verwenden.
Unsere Beispiel-Datei oben sähe dann so aus:
iso2c;year;Exporte
AT;2012;53,97
AT;2013;53,44
AT;2014;53,38Zum Glück können wir das Spaltentrennzeichen über das Argument sep und das
Kommatrennzeichen über das Argument dec manuell spezifizieren:20
daten_pfad <- here::here("data/tidy/export_daten_dt.csv")
daten <- data.table::fread(
daten_pfad, colClasses = c("character", "double", "double"),
sep = ";",
dec = ","
)
daten#> iso2c year Exporte
#> 1: AT 2012 53.97
#> 2: AT 2013 53.44
#> 3: AT 2014 53.38data.table::fread() verfügt noch über viele weitere Spezifizierungsmöglichkeiten, mit denen Sie
sich am besten im konkreten Anwendungsfall vertraut machen.
Auch ein Blick in die Hilfeseite ist recht illustrativ.
Für die meisten Anwendungsfälle sind Sie jetzt aber gut aufgestellt.
Anmerkungen zu komprimierten Dateien: Häufig werden Sie auch komprimierte Dateien einlesen wollen. Gerade komprimierte csv-Dateien kommen häufig vor. In den meisten Fällen können Sie diese Dateien direkt mit
data.table::fread()einlesen. Falls nicht, können Siedata.table::fread()aber auch dem entsprechenden UNIX-Befehl zum Entpacken als Argumentcmdübergeben, also z.B.data.table::fread("unzip -p data/gezipte_daten.csv.bz2"). Weitere Informationen finden Sie sehr einfach im Internet.
Auch wenn csv-Dateien die am weitesten verbreiteten Daten sind:
es gibt natürlich noch viele weitere Formate mit denen Sie in Kontakt kommen werden.
Hier möchte ich exemplarisch auf drei weitere Formate (.rds, .rdata und
.dta) eingehen:
R verfügt über zwei ‘hauseigene’ Formate, die sich extrem gut zum Speichern von
größeren Daten eignen, aber eben nur von R geöffnet werden können.
Diese Dateien enden mit .rds, bzw. mit .RData oder .Rda, wobei .Rda
nur eine Abkürzung für .RData ist.
Dabei gilt, dass .rds-Dateien einzelne R-Objekte enthalten, z.B. einen
einzelnen Datensatz, aber auch jedes andere Objekt (Vektor, Liste, etc.)
kann als .rds-Dateie gespeichert werden.
Solche Dateien können mit der Funktion readRDS() gelesen werden, die als
einziges Argument den Dateinamen annimmt:
daten_pfad <- here::here("data/tidy/export_daten.rds")
daten <- readRDS(daten_pfad)
daten#> Land Jahr BIP
#> 1 DEU 2011 1
#> 2 DEU 2012 2.RData-Dateien können auch mehrere Objekte enthalten.
Zudem gibt die entsprechende Funktion load() kein Objekt aus, dem Sie einen
Namen zuweisen können.
Vielmehr behalten die Objekte den Namen, mit dem sie ursprünglich gespeichert
wurden.
In diesem Fall wurden in der Datei data/tidy/test_daten.RData der Datensatz
test_dat und der Vektor test_vec gespeichert.
Entsprechend sind sie nach dem Einlesen verfügbar:
load(here::here("data/tidy/test_daten.RData"))
test_dat#> a b
#> 1 1 3
#> 2 2 4test_vec#> [1] "Test Vektor"Die Verwendung von .RData ist besonders dann hilfreich, wenn Sie mehrere
Objekte speichern wollen und wenn einige dieser Objekte keine Datensätze sind,
für die auch andere Formate zur Verfügung stehen.
Ein in der Ökonomik häufig verwendetes Format ist das von der Software
STATA verwendete Format .dta.
Um Dateien in diesem Format lesen zu können verwenden Sie die Funktion
read_dta() aus dem Paket haven (Wickham und Miller 2019),
die als einziges Argumente den Dateinamen akzeptiert:
dta_datei <- here::here("data/tidy/export_daten.dta")
dta_daten <- haven::read_dta(dta_datei)
head(dta_daten, 2)#> # A tibble: 2 x 3
#> iso2c year Exporte
#> <chr> <dbl> <dbl>
#> 1 AT 2012 54.0
#> 2 AT 2013 53.4Das Paket haven stellt auch Funktionen zum Lesen von SAS oder SPSS-Dateien bereit.
Exkurs: Große Zahlen und Probleme mit
int64Wir haben in Kapital 3 ja bereits den etwas besonderen Datentypbit64::integer64kennen gelernt. Da dieser Datentyp mit einigen Operatoren inkompatibel ist und merkwürdiges Verhalten verursacht wenn das Paketbit64nicht installiert ist, sollten wir seine Verwendung unbedingt vermeiden. Wenn Sie aber mitdata.table::freadeinen Datensatz einlesen, der ganze Zahlen beinhaltet, die größer sind als 2147483647, dann werden diese automatisch alsbit64::integer64kodiert.21
large_nb_frame <- data.table::fread(
here::here("data/tidy/BIPKonsum.csv"))
str(large_nb_frame, vec.len=3)#> Classes 'data.table' and 'data.frame': 27 obs. of 3 variables:
#> $ Jahr : int 1992 1993 1994 1995 1996 1997 1998 1999 ...
#> $ Konsumausgaben:integer64 1601342020000 1611040070000 1647698700000 1681059990000 1706080960000 1719658230000 ...
#> $ BIP :integer64 2077722320000 2057855860000 2108425030000 2145061880000 2162606290000 2202597220000 ...
#> - attr(*, ".internal.selfref")=<externalptr>Das können wir verhindern, indem wir die Spaltentypen explizit über
colClassesspezifizieren oder aber zur Sicherheit das Argumentinteger64auf"double"setzen:
large_nb_frame <- data.table::fread(
here::here("data/tidy/BIPKonsum.csv"),
integer64 = "double")
str(large_nb_frame, vec.len=3)#> Classes 'data.table' and 'data.frame': 27 obs. of 3 variables:
#> $ Jahr : int 1992 1993 1994 1995 1996 1997 1998 1999 ...
#> $ Konsumausgaben: num 1.60e+12 1.61e+12 1.65e+12 1.68e+12 ...
#> $ BIP : num 2.08e+12 2.06e+12 2.11e+12 2.15e+12 ...
#> - attr(*, ".internal.selfref")=<externalptr>4.3.2 Speichern von Daten
Im Vergleich zum Einlesen von Daten ist das Speichern deutlich einfacher, weil sich die Daten ja bereits in einem vernünftigen Format befinden. Die größte Frage hier ist also: in welchem Dateiformat sollten Sie Ihre Daten speichern?
In der großen Mehrheit der Fälle ist diese Frage klar mit .csv zu beantworten.
Dieses Format ist einfach zu lesen und absolut plattformkompatibel.
Es hat auch nicht die schlechtesten Eigenschaften was Lese- und Schreibgeschwindigkeit
angeht, insbesondere wenn man die Daten komprimiert.
Die schnellste und meines Erachtens mit Abstand beste Funktion zum Schreiben von
csv-Dateien ist die Funktion fwrite() aus dem Paket data.table.
Angenommen wir haben einen Datensatz test_data, den wir im Unterordner
data/tidy als test_data.csv speichern wollen.
Das geht mit data.table::fwrite() ganz einfach:
datei_name <- here::here("data/tidy/test_data.csv")
data.table::fwrite(test_data, file = datei_name)Neben dem zu schreibenden Objekt als erstem Argument benötigen Sie noch das
Argument file, welches den Namen und Pfad der zu schreibenden Datei spezifiziert.
Der Übersicht halber ist es oft empfehlenswert diesen Pfad zuerst als
character-Objekt zu speichern und dann an die Funktion data.table::fwrite() zu übergeben.
data.table::fwrite() akzeptiert noch einige weitere optionale Argumente, die Sie im
Großteil der Fälle aber nicht benötigen.
Schauen Sie bei Interesse einfach einmal in die Hilfefunktion!
Falls Ihr Datensatz im csv-Format doch zu groß ist, Sie aber aufgrund von
Kompatibilitätsanforderungen kein spezialisiertes Format benutzen wollen, bietet
es sich an die csv-Datei zu komprimieren.
Natürlich könnten Sie das händisch in Ihrem Datei-Explorer machen, aber das ist vollkommen überholt.
Sie können das gleich in R miterledigen indem Sie z.B. die Funktion gzip()
aus dem Paket R.utils (Bengtsson 2019) verwenden:
csv_datei_name <- here::here("data/tidy/test_data.csv")
data.table::fwrite(test_data, file = csv_datei_name)
R.utils::gzip(
csv_datei_name,
destname=paste0(csv_datei_name, ".gz"),
overwrite = TRUE, remove=TRUE)Diese Funktion akzeptiert als erstes Argument den Pfad zu der zu komprimierenden
Datei, also zweites Argument (destname) den Namen, den die komprimierte Datei
tragen soll und einige weitere optionale Argumente. Häufig bietet sich
overwrite = TRUE an, um alte Versionen der komprimierten Datei im Zweifel zu
überschreiben, und remove=TRUE um die un-komprimierte Datei nach erfolgter
Komprimierung zu löschen.
Hinweise zu verschiedenen zip-Formaten: Die Funktion
R.utils::gzip()komprimiert eine Datei mit dem GNU zip Algorithmus. Die resultierende komprimierten Dateien sollten mit der zusätzlichen Endung.gzgekennzeichnet werden.R.utils::gzip()ist eine relativ schnell arbeitende Funktion, allerdings mit mäßigen Kompressionseigenschaften. Wenn Sie bereit sind längere Arbeitszeit für ein besseres Kompressionsergebnis in Kauf zu nehmen, sollten Sie sich die FunktionR.utils::bzip2()ansehen, welche den bzip2-Algorithmus implementiert. Dieser hat eine deutlich bessere Kompressionsrate (die komprimierten Dateien sind also deutlich kleiner), allerdings istR.utils::bzip2()auch deutlich langsamer alsR.utils::gzip(). Dateien, die mitR.utils::bzip2()komprimiert wurden, sollten mit der Endung.bz2gekennzeichnet werden. Entsprechend sieht der Code von oben mitR.utils::bzip2()anstattR.utils::gzip()folgendermaßen aus:
csv_datei_name <- here::here("data/tidy/test_data.csv")
data.table::fwrite(test_data, csv_datei_name)
R.utils::bzip2(
csv_datei_name,
destname=paste0(csv_datei_name, ".bz2"),
overwrite = TRUE)Einen Vergleich der Kompressionseigenschaften und Lese- und Schreibgeschwindigkeiten ist immer auch kontextabhängig, im Internet finden sich viele Diskussionen zu dem Thema. Am Anfang sind Sie mit
R.utils::gzip()undR.utils::bzip2()aber eigentlich für alle relevanten Fälle gut aufgestellt.
Die oben bereits vorgestellten R-spezifischen Formate .Rdata und .rds verfügen
über deutliche Geschwindigkeits- und Komprimierungsvorteile gegenüber dem csv-Format
und sind dabei trotzdem vollkommen plattformkompatibel.
Einziger Nachteil:
alle Irren, die nicht R benutzen, können Ihre Daten nicht öffnen.
Manchmal mag das eine verdiente Strafe, manchmal aber auch ein Ausschlusskriterium
sein.
saveRDS(object = test_data, file = here::here("data/tidy/export_daten.rds"))Wie Sie sehen sind zwei Argumente zentral: das erste Argument, object
spezifiziert das zu speichernde Objekt und file den Dateipfad.
Darüber hinaus können Sie mit dem optionalen Argument compress hier die
Kompressionsart auswählen. Ähnlich wie oben gilt, dass gz am schnellsten und
bz am stärksten ist. xz liegt in der Mitte.
Wenn Sie mehrere Objekte auf einmal speichern möchten können Sie das über das
Format .RData machen.
Die entsprechende Funktion ist save().
Zwar können Sie einfach alle zu speichernden Objekte als die ersten
Argumente an die Funktion übergeben, es ist aber übersichtlicher das
über das Argument list zu erledigen.
Der folgende Code speichert die beiden Objekte test_data und daten in der
Datei "data/tidy/datensammlung.Rdata":
save(
list=c("test_data", "daten"),
file=here::here("data/tidy/datensammlung.RData")
)Wie saveRDS() können Sie bei save() über das Argument compress den
Kompressionsalgorithmus auswählen,
allerdings können Sie mit compression_level zusätzlich noch die Stärke von 1
(schnell, aber wenig Kompression) bis 9 (langsamer, aber starke Kompression)
auswählen.
Da, wie oben erwähnt, gerade in der Ökonomik auch häufig mit der kostenpflichtigen Software
STATA gearbeitet wird, möchte ich noch
kurz erkläutern, wie man einen Datensatz im STATA-Format .dta speichern kann.
Dazu verwenden wir die Funktion write_dta()
aus dem Paket haven.
haven::write_dta(
test_data, here::here("data/tidy/test_daten.dta"))Für SAS- und SPSS-Daten gibt es ähnliche Funktionen, die ebenfalls durch das
haven-Paket bereitgestellt werden.
Hinweis: Gerade bei großen Datensätzen kommt es wirklich sehr auf die Lese- und Schreibgeschwindigkeit von Funktionen an. Auch stellt sich hier die Frage nach dem besten Dateiformat noch einmal viel deutlicher als das bei kleinen Datensätzen der Fall ist und sich die Formatfrage vor allem um das Thema ‘Kompatibilität’ dreht. Einige nette Beiträge, die verschiedene Funktionen und Formate bezüglich ihrer Geschwindigkeit vergleichen finden Sie z.B. hier oder
hier.
4.4 Verarbeitung von Daten (‘data wrangling’)
Nachdem Sie Ihre Daten erhoben haben, müssen Sie die Rohdaten in eine Form bringen, mit der Sie sinnvoll weiterarbeiten können. Dieser Prozess wird oft als ‘Datenaufbereitung’ bezeichnet und stellt häufig einen der zeitaufwendigsten Arbeitsschritte in der Forschungsarbeit dar: Laut dieser Umfrage macht es sogar 60 % der Arbeitszeit von Datenspezialist*innen aus. Entsprechend wichtig ist es, sich mit den typischen Arbeitsschritten und Algorithmen vertraut zu machen um in diesem aufwendigen Arbeitsschritt Zeit zu sparen.
Ein großes Problem in der Forschungspraxis ist häufig, dass Forscher*innen den Datenaufbereitungsprozess nicht richtig dokumentieren. In diesem Fall ist unklar was für Änderungen an den Rohdaten vorgenommen wurden bevor die eigentliche Analyse begonnen wurde. Das führt zu unreproduzierbarer und intransparenter Forschung. Daher ist es wichtig, alle Änderungen, die Sie im Rahmen der Datenaufbereitung vornehmen zu dokumentieren.
Am einfachsten ist es, für die Datenaufbereitung einfach ein R-Skript zu schreiben,
in dem Sie die Rohdaten einlesen und am Ende die aufbereiteten Daten unter neuem
Namen speichern.
Am besten legen Sie in Ihrem Ordner data zwei Unterordner an:
Die Rohdaten speichern Sie dann in einem Unterordner raw, die bearbeiteten und
aufbereiteten Daten in einem Unterordner tidy.
So behalten Sie immer den Überblick.
Nie sollten Sie Ihre Rohdaten überschreiben!
Damit sind Sie in Ihrer Forschung vollkommen transparent und es entsteht Ihnen
im Prinzip keine Mehrarbeit (siehe Abbildung 4.4).

Abbildung 4.4: Eine übersichtliche Art und Weise Ihre Daten zu speichern. Die Dateien im Unterordner raw werden nie geändert. Die Datei exports-tidy im Ordner tidy wurde mit dem Skript exports-preparation.R aus dem Rohdatensatz exports-raw.csv erstellt.
In diesem Abschnitt lernen Sie Lösungen für Probleme, die typischerweise während der Datenaufbereitung auftreten. Dafür beschäftigen wir uns zunächst mit dem gewünschten Ergebnis: sogenannter tidy data. Diese Art von Datensätzen sollte das Ergebnis jeder Datenaufbereitung sein.
Auf dem Weg zu tidy data bedarf es häufig einer Transformation von langen und breiten Datensätzen. Außerdem werden Sie häufig mehrere Datensätze zusammenführen und Ihre Daten filtern, selektieren und aggregieren. Zudem möchten Sie manchmal Daten auch reduzieren und zusammenfassen.
Beispiel für berühmte Menschen mit miserabler Datenaufbereitung: Der Reinhart-Rogoff Skandal
Eines der dramatischsten Beispiele für Fehler in der Datenaufbereitung mit katastrophalen realweltlichen Implikationen ist der Reinhart-Rogoff-Skandal. Carmen Reinhart und Kenneth Rogoff haben in ihrem einflussreichen Paper Growth in a Time of Debt einen negativen Effekt von übermäßiger Staatsverschuldung auf wirtschaftliches Wachstum festgestellt. Als der PhD-Student Thomas Herndon während eines Seminars das Paper replizieren sollte, bekam er Probleme. Dankenswerterweise sendete ihm Carmen Reinhard den Datensatz zu, allerdings stellte sich heraus, dass durch einen Excel-Fehler einige Länder aus der Stichprobe gefallen waren. Mit der kompletten Stichprobe löste sich der im ursprünglichen Paper identifizierte Zusammenhang auf (Herndon, Ash, und Pollin 2013). Das ist besonders dramatisch, da das Paper nicht nur zahlreiche Preise gewonnen hat, sondern auch als wichtige Begründung für die in Europa implementierte Austeritätspolitik fungierte. Klar ist: wäre der Datenaufbereitungsprozess transparent und offen durchgeführt und dokumentiert worden, wäre der Fehler wahrscheinlich deutlich einfacher und früher gefunden worden.
4.4.1 Das Konzept von ‘tidy data’
Die Rohdatensätze, die wir erheben oder aus dem Internet herunterladen haben
oft eine abenteuerliche Form und wir können in der Regel nicht direkt mit der
statistischen Analyse anfangen.
Die meisten Statistik-Pakete und Funktionen setzen eine bestimmte ‘aufgeräumte’
Form der Daten voraus. Wickham (2014) beschreibt diese Form als tidy data22 und es ist unser Ziel durch die Datenaufbereitung die
verschiedenen Rohdatensätze in tidy data zu verwandeln. Die daraus resultierenden
Datensätze können dann separat gespeichert werden, damit wir die Datenaufbereitung
nicht jedes Mal erneut durchführen müssen (im Abschnitt
Abschließende Bemerkungen wird ein entsprechender
Vorschlag für eine hilfreiche Ordnerstruktur beschrieben).
Aber was zeichnet tidy data aus? Wie von Wickham (2014) beschrieben kann ein Datensatz
auf vielerlei Art und Weise ‘unordentlich’ sein, aber nur auf eine Art und Weise
‘tidy’. Ein ‘tidy’ Datensatz ist durch folgende drei Eigenschaften gekennzeichnet:
- Jede Spalte korrespondiert zu genau einer Variable
- Jede Zeile korrespondiert zu genau einer Beobachtung
- Jede Zelle korrespondiert zu einem einzelnen Wert
Punkt (1) verlangt, dass jede Spalte zu einer Variable korrespondiert und es keine
Spalten gibt, die zu keiner Variable korrespondieren.
Wenn wir also Daten zum BIP in verschiedenen Ländern über die Zeit erheben
impliziert das, dass wir es mit drei Variablen zu tun haben: dem Land, dem
Jahr und dem BIP. Entsprechend sollte unser Datensatze genau drei Spalten
haben, die jeweils zu diesen Variablen korrespondieren.
Punkt (2) verlangt, dass jede Zeile zu genau einer Beobachtung korrespondiert. In unserem Beispiel sollte also jede Zeile zu der Beobachtung des BIP in genau einem Land zu genau einem Zeitraum korrespondieren - und z.B. nicht die Beobachtungen für ein einziges Land zu allen möglichen Zeiträumen sammenln.
Punkt (3) ist meistens in unseren Anwendungsfällen ohnehin erfüllt.
Er verlangt, dass jede Zelle in unserem Datensatz genau einen Wert enthält, und
z.B. nicht nochmal eine Liste mit mehreren Werten, wie es ja bei einem
data.frame auch
möglich wäre.
Beispiel ‘tidy data’: Der folgende Datensatz ist ‘tidy’ im gerade beschriebenen Sinn:
#> Land Jahr Exporte Arbeitslosigkeit
#> 1: AT 2013 53.44129 5.335
#> 2: AT 2014 53.38658 5.620
#> 3: DE 2013 45.39788 5.231
#> 4: DE 2014 45.64482 4.981Wir haben vier Spalten, die jeweils zu einer der vier Variablen
Land,Jahr,ExporteundArbeitslosigkeitkorrespondieren. Jede Zeile korrespondiert zur Beobachtung vonBIPundExportein genau einem Jahr in genau einem Land. Und die einzelnen Zellen enthalten genau einen Wert, jeweils für das Land, das Jahr, die Exporte und die Arbeitslosigkeit.
Beispiel: Verstoß gegen (1) : Der folgende Datensatz, welcher nur Informationen zu den Exporten und für das Jahr 2014 enthält, ist nicht ‘tidy’, da er gegen Anforderung (1) verstößt:
#> Land Variable 2014
#> 1 AT Exporte 53.38658
#> 2 DE Exporte 45.64482Hier haben wir drei Variablen,
Land,JahrundExporte, die im Jahr 2014 erhoben wurden. In der SpalteVariablekommen aber die ersten drei Variablen vor und die Spalte2013korrespondiert zu einer Ausprägung der VariableJahr, aber nicht zu den eigentlichen Variablen als solchen. Die Bedeutung dieser Unterscheidung wird im nächsten Beispiel deutlich.
Beispiel: Verstoß gegen (1) und (2): Wenn wir in dem ersten Datensatz alle Informationen belassen, würde er in der gerade dargestellten Form sowohl gegen (1) als auch (2) verstoßen:
#> Land Variable 2013 2014
#> 1 AT Arbeitslosigkeit 5.33500 5.62000
#> 2 AT Exporte 53.44129 53.38658
#> 3 DE Arbeitslosigkeit 5.23100 4.98100
#> 4 DE Exporte 45.39788 45.64482Jetzt ist nicht nur die Anforderung, dass jede Spalte zu einer Variable korrespondiert, verletzt, sondern auch die Anforderung, dass jede Zeile zu genau einer Beobachtung korrespondiert, da wir wegen der zwei Jahre in jeder Zeile zwei Beobachtungen haben. Ebenfalls sehr häufig kommt folgendes Format vor, das ebenfalls (1) und (2) widerspricht:
#> Land Jahr Variable Wert
#> 1 AT 2013 Exporte 53.44129
#> 2 AT 2014 Exporte 53.38658
#> 3 DE 2013 Exporte 45.39788
#> 4 DE 2014 Exporte 45.64482
#> 5 AT 2013 Arbeitslosigkeit 5.33500
#> 6 AT 2014 Arbeitslosigkeit 5.62000
#> 7 DE 2013 Arbeitslosigkeit 5.23100
#> 8 DE 2014 Arbeitslosigkeit 4.98100Beispiel: Verstoß gegen (3) Verstöße gegen die dritte Anforderung kommen in der Praxis in der Regel seltener vor, sind aber auch unschön:
d <- data.frame(Land=c("DE", "AT"))
d[["Wichtige Industrien"]] <- list(
c("Autos", "Medikamente"), c("Stahlproduktion", "Holz"))
d#> Land Wichtige Industrien
#> 1 DE Autos, Medikamente
#> 2 AT Stahlproduktion, Holz4.4.2 Von langen und breiten Datensätzen
Die Datenaufbereitung umfasst häufig das Wechseln zwischen der so genannten ‘langen’ (oder ‘gestapelten’) und ‘breiten’ (‘ungestapelten’) Datenform. Die erste ist für die statistische Verarbeitung, die zweite für das menschliche Auge besser geeignet.
‘Lange’ Daten haben in der Regel viele Zeilen und wenige Spalten.
Alle tidy Datensätze sind im langen Datenformat. ‘Breite’ Daten haben
mehr Spalten und weniger Zeilen und sind häufig das, was wir aus dem
Internet herunterladen. Im Folgenden ist der gleiche Datensatz einmal im
langen und einmal im breiten Format dargestellt.
Zuerst das ‘lange’ Format, in dem wir verhältnismäßig viele Zeilen haben:
#> Land Jahr Exporte
#> 1: AT 2013 53.44129
#> 2: AT 2014 53.38658
#> 3: DE 2013 45.39788
#> 4: DE 2014 45.64482Und hier das ‘breite’ Format mit verhältnismäßig mehr Spalten:
#> Land Variable 2013 2014
#> 1 AT Exporte 53.44129 53.38658
#> 2 DE Exporte 45.39788 45.64482Häufig werden Sie während Ihrer Datenaufbereitung mehrmals zwischen den beiden Formaten hin und her wechseln, da für manche statistischen Zwischenschritte das eine, für andere das andere Format besser ist.23
Um zwischen den Formaten hin und herzuwechseln verwenden wir vor allem die
Funktionen pivot_longer() und pivot_wider() aus dem Paket
tidyr (Wickham und Henry 2019), welches auch Teil des
tidyverse ist.24
Wir verwenden tidyr::pivot_longer() um einen Datensatz ‘länger’ zu machen.
Wir verwenden dazu folgenden Datensatz als Ausgangsbeispiel, der Werte
für die Arbeitslosigkeit in Deutschland und Österreich in zwei Jahren
enthält:
data_wide#> Land 2013 2014
#> 1 AT 5.335 5.620
#> 2 DE 5.231 4.981Das erste Argument für tidyr::pivot_longer() heißt data und nimmt den Datensatz,
den wir länger machen wollen. In unserem Beispiel also data_wide.
Das zweite Argument heißt cols und beschreibt die Spalten an denen Änderungen
vorgenommen werden sollen.
In unserem Falle sind das die Spalten 2013 und 2014.
Um hier eine Liste von Spaltennamen zu übergeben verwenden wir die Hilfsfunktion
any_of(), die es uns erlaubt die Spaltennamen als character zu schreiben.
Das Argument wird also als cols=any_of(c("2013", "2014")) spezifiziert.
Das dritte Argument, names_to akzeptiert einen character, der den Namen der
neu zu schaffenden Spalte beschreibt.
In unserem Fall macht es Sinn, diese Spalte Jahr zu nennen.
Das vierte Argument, values_to spezifiziert den Namen der Spalte, welche die
Werte des verlängerten Datensatzes beschreibt.
In unserem Falle bietet sich der Name Arbeitslosenquote an, da es sich bei dem
Datensatz um Arbeitslosenquotenstatistiken handelt.
Insgesamt erhalten wir damit den folgenden Funktionsaufruf:
data_long <- tidyr::pivot_longer(
data = data_wide,
cols = any_of(c("2013", "2014")),
names_to = "Jahr",
values_to = "Arbeitslosenquote"
)
data_long#> # A tibble: 4 x 3
#> Land Jahr Arbeitslosenquote
#> <chr> <chr> <dbl>
#> 1 AT 2013 5.34
#> 2 AT 2014 5.62
#> 3 DE 2013 5.23
#> 4 DE 2014 4.98Wenn wir den umgekehrten Weg gehen wollen, also einen langen Datensatz ‘breiter’
machen wollen, verwenden wir die Funktion tidyr::pivot_wider().
Hier wird die Anzahl der Zeilen reduziert und die Anzahl der Spalten erhöht
Gehen wir einmal vom gerade produzierten Datensatz aus:
data_long#> # A tibble: 4 x 3
#> Land Jahr Arbeitslosenquote
#> <chr> <chr> <dbl>
#> 1 AT 2013 5.34
#> 2 AT 2014 5.62
#> 3 DE 2013 5.23
#> 4 DE 2014 4.98Die Funktion tidyr::pivot_wider() verlangt als erstes Argumt wieder data, also
den zu manipulierenden Datensatz. Im Beispiel ist das data_long.
Das zweite Argument, id_cols, legt die Spalten fest, die nicht verändert
werden sollen, weil sie die Beobachtung als solche spezifizieren. In unserem
Fall ist das die Spalte Land, aber manchmal ist das auch mehr als eine Spalte.
In dem Fall ist die Verwendung der Funktion any_of() wie im Beispiel oben
notwendig, im Falle von einer Spalte wie hier ist das optional.
Das dritte Argument, names_from verlangt nach den Spalten, deren Inhalte im
breiten Datensatz als einzelne Spalten aufgeteilt werden sollen.
In unserem Falle wäre das die Spalte Jahr, weil wir in unserem breiten
Datensatz separate Spalten für die einzelnen Jahre haben wollen.
Das vierte Argument ist values_from und spezifiziert die Spalte aus der die
Werte für die neuen Spalten genommen werden sollen.
In unserem Falle wäre das die Spalte Arbeitslosenquote, da wir ja in die
Spalten für die einzelnen Jahre die Arbeitslosenquoten schreiben wollen.
Insgesamt sieht der Funktionsaufruf also so aus:
data_wide_neu <- tidyr::pivot_wider(
data = data_long,
id_cols = any_of("Land"),
names_from = "Jahr",
values_from = "Arbeitslosenquote"
)
data_wide_neu#> # A tibble: 2 x 3
#> Land `2013` `2014`
#> <chr> <dbl> <dbl>
#> 1 AT 5.34 5.62
#> 2 DE 5.23 4.98Zum Schluss möchten wir uns noch ein Beispiel ansehen in dem wir beide Befehle nacheinander verwenden. Betrachten wir folgenden Datensatz, der Beobachtungen sowohl zur Arbeitslosenquote also auch zu den Exporten enthält:
#> # A tibble: 4 x 5
#> Land Variable `2012` `2013` `2014`
#> <chr> <chr> <dbl> <dbl> <dbl>
#> 1 AT Exporte 54.0 53.4 53.4
#> 2 AT Arbeitslosigkeit 4.86 5.34 5.62
#> 3 DE Exporte 46.0 45.4 45.6
#> 4 DE Arbeitslosigkeit 5.38 5.23 4.98Eine tidy Version dieses Datensatzes sähe so aus:
#> # A tibble: 6 x 4
#> Land Jahr Exporte Arbeitslosigkeit
#> <chr> <chr> <dbl> <dbl>
#> 1 AT 2012 54.0 4.86
#> 2 AT 2013 53.4 5.34
#> 3 AT 2014 53.4 5.62
#> 4 DE 2012 46.0 5.38
#> 5 DE 2013 45.4 5.23
#> 6 DE 2014 45.6 4.98Leider ist diese Transformation nicht in einem Schritt zu machen.
Als erstes müssen wir nämlich den Datensatz länger machen, indem die Jahre
in ihre eigene Spalte gepackt werden, und dann muss der Datensatz breiter
gemacht werden indem die Variablen Exporte und Arbeitslosigkeit ihre
eigene Spalte bekommen.
Wir machen uns dabei zu Nutze, dass wir dem Argument cols auch die Namen der
Spalten geben können, die wir nicht transformieren wollen. Dazu stellen wir
der Liste der Namen ein - voran und R wird entsprechend alle hier nicht
genannten Spalten für die Transformation verwenden:
data_al_exp_longer <- tidyr::pivot_longer(
data = data_al_exp,
cols = -any_of(c("Land", "Variable")),
names_to = "Jahr",
values_to = "Wert"
)
head(data_al_exp_longer, 2)#> # A tibble: 2 x 4
#> Land Variable Jahr Wert
#> <chr> <chr> <chr> <dbl>
#> 1 AT Exporte 2012 54.0
#> 2 AT Exporte 2013 53.4Beachten Sie wie wir diesmal das Argument cols spezifiziert haben:
anstatt alle Jahre in die Funktion any_of() zu schreiben, haben wir stattdessen
die Spalten spezifiziert, die nicht bearbeitet werden sollen und das mit einem
- vor any_of() gekennzeichnet.
Das ist vor allem dann hilfreich wenn wir sehr viele Spalten zusammenfassen
wollen, was häufig vorkommt, wenn es sich bei den Spalten um Jahre handelt.
Um unser gewünschtes Endergebnis zu erhalten müssen wir diesen Datensatz nun nur noch breiter machen:
data_al_exp_tidy <- tidyr::pivot_wider(
data = data_al_exp_longer,
id_cols = any_of(c("Land", "Jahr")),
values_from = "Wert",
names_from = "Variable"
)
data_al_exp_tidy#> # A tibble: 6 x 4
#> Land Jahr Exporte Arbeitslosigkeit
#> <chr> <chr> <dbl> <dbl>
#> 1 AT 2012 54.0 4.86
#> 2 AT 2013 53.4 5.34
#> 3 AT 2014 53.4 5.62
#> 4 DE 2012 46.0 5.38
#> 5 DE 2013 45.4 5.23
#> 6 DE 2014 45.6 4.98Insgesamt sähe der Code damit folgendermaßen aus:
data_al_exp_longer <- tidyr::pivot_longer(
data = data_al_exp,
cols = -any_of(c("Land", "Variable")),
names_to = "Jahr",
values_to = "Wert")
data_al_exp_tidy <- tidyr::pivot_wider(
data = data_al_exp_longer,
id_cols = any_of(c("Land", "Jahr")),
values_from = "Wert",
names_from = "Variable")Da die Kombination solcher Schritte in der Praxis sehr häufig vorkommt und
man die vielen Zuweisungen der Übersicht halber vermeiden möchte, bieten die
Pakete des tidyverse eine schöne Möglichkeit, den Code zu verkürzen:
die so genannte Pipe %>%.
Mit %>% geben Sie ein Objekt direkt an die nächste Funktion weiter.
Dort wird das Ergebnis des vorherigen Aufrufs automatisch als erstes Argument
verwendet.
Wir könnten also auch schreiben:
data_al_exp_tidy <- data_al_exp %>%
tidyr::pivot_longer(
cols = -any_of(c("Land", "Variable")),
names_to = "Jahr",
values_to = "Wert") %>%
tidyr::pivot_wider(
id_cols = any_of(c("Land", "Jahr")),
values_from = "Wert",
names_from = "Variable")Das ist gleich viel besser lesbar!
In der ersten Zeile schreiben wir nur das Ausgangsobjekt data_al_exp,
welches über %>% dann unmittelbar als erstes Argument an tidyr::pivot_longer()
übergeben wird. Da es sich beim ersten Argument um data handelt ist das genau
das was wir wollen.
Das Schreiben mit %>% führt in der Regel zu sehr transparentem und
nachvollziehbarem Code, da Sie die einzelnen Manipulationsschritte schön von
oben nach unten nachlesen können.
Tipp: Streng genommen gibt
%>%den Output der aktuellen Zeile nicht automatisch als erstes Argument für den Funktionsaufruf der nächsten Zeile weiter. Das ist nur das Standardverfahren. Eigentlich gibt es den Output als.weiter. Das erlaubt Ihnen den Output auch in einem anderen als dem ersten Argument zu verwenden.
Wir könnten also auch expliziter schreiben:
data_al_exp_tidy <- data_al_exp %>%
tidyr::pivot_longer(
data = .,
cols = -any_of(c("Land", "Variable")),
names_to = "Jahr",
values_to = "Wert") %>%
tidyr::pivot_wider(
data = .,
id_cols = any_of(c("Land", "Jahr")),
values_from = "Wert",
names_from = "Variable")Das ist hilfreich, wenn Sie den Output einer Zeile nicht als erstes, sondern z.B. als zweites Argument in der nächsten Funktion verwenden wollen. Dann verwenden Sie den
.einfach explizit da wo Sie ihn brauchen. Da Sie die Argumente ja nicht in der richtigen Reihenfolge angeben müssen solange die Namen stimmen funktioniert also auch folgender Code:
data_al_exp_tidy <- data_al_exp %>%
tidyr::pivot_longer(
cols = -any_of(c("Land", "Variable")),
names_to = "Jahr",
values_to = "Wert",
data = .) %>%
tidyr::pivot_wider(
id_cols = any_of(c("Land", "Jahr")),
values_from = "Wert",
names_from = "Variable",
data = .)Beide Funktionen, tidyr::pivot_wider() and tidyr::pivot_longer(), können noch viel
komplexere Probleme lösen.
Für weitere Anwendungen verweisen wir auf die offizielle
Dokumentation.
4.4.3 Zusammenführen von Daten
Häufig möchten Sie mehrere Datensätze zusammenführen. Nehmen wir an, Sie hätten einen Datensatz, der Informationen über das BIP in verschiedenen Ländern über die Zeit enthält, und einen zweiten Datensatz, der Informationen über die Einkommensungleichheit in ähnlichen Ländern enthält.
#> Jahr Land BIP
#> 1 2010 DEU 1
#> 2 2011 DEU 2
#> 3 2012 DEU 3
#> 4 2010 AUT 4
#> 5 2011 AUT 5
#> 6 2012 AUT 6#> year country Gini
#> 1 2010 DEU 1
#> 2 2011 DEU 2
#> 3 2012 AUT 3
#> 4 2013 AUT 4Um den Zusammenhang zwischen Einkommensungleichheit und BIP zu untersuchen, möchten Sie die Datensätze zusammenführen, und dabei die Länder und Jahre richtig kombinieren.
Zum Glück hat das Paket dplyr, das ein Teil des tidyverse darstellt, für jede
Situation die passende Funktion parat.
Insgesamt gibt es im Paket die folgenden Funktionen, die alle dafür verwendet
werden können, zwei Datensätze zusammenzuführen:
inner_join(), left_join(), right_join(), full_join(), semi_join(),
nest_join() und anti_join().
Wir vergleichen nun das Verhalten der verschiedenen Funktionen mit Hilfe der beiden Beispiel-Datensätze zum BIP und zur Ungleichheit und fassen sie am Ende des Abschnitts nochmals in einer Tabelle (siehe Tabelle 4.3) zusammen.
Wie alle Funktionen der *_join()-Familie aus dem dplyr Paket verlangt left_join() zwei
notwendige Argumente, x und y, welche die beiden zu verbindenden Datensätze
spezifizieren.
Wir nennen dabei x den ‘linken’ und y den ‘rechten’ Datensatz.
Die Funktion dplyr::left_join() sollten Sie verwenden, wenn Sie zu allen Zeilen in x
(dem ‘linken’ Datensatz) die passenden Werte aus y hinzufügen wollen.
Wenn eine Beobachtung nur in y vorkommt, wird diese im finalen Datensatz nicht
berücksichtigt.
Wenn eine Beobachtung nur in x vorkommt, wird in den Spalten aus y der Wert
NA eingefügt.
Man könnte sagen, der ‘linke’ Datensatz hat in dplyr::left_join() ‘Priorität’.
Um zu spezifizieren gemäß welcher Spalten die Datensätze verbunden werden sollen
können wir über das optionale Argument id die ‘ID-Spalten’ definieren.
Diese Spalten identifizieren eine gemeinsame Beobachtung in x und y.
In unserem Beispiel von oben wären das die Spalten Jahr (in data_bip) und
year (in data_gini) sowie Land (in data_bip) und country (in data_gini).
Um die Datensätze so zu kombinieren, dass wir die Daten den Ländern und
Jahren entsprechend zusammenführen schreiben wir:
by=c("Jahr"="year", "Land"="country"), also den Spaltennamen in x auf die
linke Seite von = und das Pendant in y auf der rechten Seite vom =.
Im Falle von dplyr::left_join() ergibt sich also:
data_bip_gini_left_join <- dplyr::left_join(
data_BIP, data_gini, by=c("Jahr"="year", "Land"="country"))
data_bip_gini_left_join#> Jahr Land BIP Gini
#> 1 2010 DEU 1 1
#> 2 2011 DEU 2 2
#> 3 2012 DEU 3 NA
#> 4 2010 AUT 4 NA
#> 5 2011 AUT 5 NA
#> 6 2012 AUT 6 3Verwenden wir dagegen data_gini als ‘linken’ und data_BIP als ‘rechten’ Datensatz
gibt dplyr::left_join() einen kürzeren gemeinsamen Datensatz aus, da es nur die
Beobachtungen aus dem rechten Datensatz übernimmt, für die es ein Pendant im
linken Datensatz gibt.
data_gini_bip_left_join <- dplyr::left_join(
data_gini, data_BIP, by=c("year"="Jahr", "country"="Land"))
data_gini_bip_left_join#> year country Gini BIP
#> 1 2010 DEU 1 1
#> 2 2011 DEU 2 2
#> 3 2012 AUT 3 6
#> 4 2013 AUT 4 NADie Funktion dplyr::inner_join() unterscheidet sich von dplyr::left_join() darin, dass
nur die Zeilen in den gemeinsamen Datensatz übernommen werden, die sowohl in x als
auch in y enthalten sind:
data_bip_gini_inner_join <- dplyr::inner_join(
data_BIP, data_gini, by=c("Jahr"="year", "Land"="country"))
data_bip_gini_inner_join#> Jahr Land BIP Gini
#> 1 2010 DEU 1 1
#> 2 2011 DEU 2 2
#> 3 2012 AUT 6 3Das Verhalten von dplyr::right_join() ist analog zu dplyr::left_join(), nur hat hier der
‘rechte’ Datensatz, also der dem Argument y übergebene Datensatz Priorität:
data_gini_bip_right_join <- dplyr::right_join(
data_gini, data_BIP, by=c("year"="Jahr", "country"="Land"))
data_gini_bip_right_join#> year country Gini BIP
#> 1 2010 DEU 1 1
#> 2 2011 DEU 2 2
#> 3 2012 AUT 3 6
#> 4 2012 DEU NA 3
#> 5 2010 AUT NA 4
#> 6 2011 AUT NA 5Wenn Sie keinem der beiden Datensätze eine Priorität einräumen möchten und
alle Zeilen in jedem Fall behalten wollen, dann wählen Sie am besten die Funktion
dplyr::full_join():
data_bip_gini_full_join <- dplyr::full_join(
data_BIP, data_gini, by=c("Jahr"="year", "Land"="country"))
data_bip_gini_full_join#> Jahr Land BIP Gini
#> 1 2010 DEU 1 1
#> 2 2011 DEU 2 2
#> 3 2012 DEU 3 NA
#> 4 2010 AUT 4 NA
#> 5 2011 AUT 5 NA
#> 6 2012 AUT 6 3
#> 7 2013 AUT NA 4Die Funktionen dplyr::semi_join() und dplyr::anti_join() funktionieren ein
wenig anders als die bisher vorgestellten Funktionen, da sie Datensätze strikt
genommen nicht zusammenführen.
Vielmehr filtern sie die Zeilen von x gemäß der in y vorkommenden Werte.
dplyr::semi_join() produziert einen Datensatz, der alle Spalten und Zeilen von x
enthält, für die es auch in y einen entsprechenden Wert gibt.
Der resultierende Datensatz enthält aber nur die Spalten vom linken Datensatz
(x). Das kann hilfreich sein, wenn der Datensatz x deutlich kleiner ist und
Sie in x keinerlei fehlende Werte haben wollen:
data_bip_gini_semi_join <- dplyr::semi_join(
data_BIP, data_gini, by=c("Jahr"="year", "Land"="country"))
data_bip_gini_semi_join#> Jahr Land BIP
#> 1 2010 DEU 1
#> 2 2011 DEU 2
#> 3 2012 AUT 6dplyr::anti_join() ist quasi das ‘Spiegelbild’ zu dplyr::semi_join():
genau wie semi_join() produziert es einen Datensatz, der nur die Spalten von
x enthält, und zwar diese, für die es in y keinen entsprechenden Wert
gibt:
data_bip_gini_anti_join <- dplyr::anti_join(
data_BIP, data_gini, by=c("Jahr"="year", "Land"="country"))
data_bip_gini_anti_join#> Jahr Land BIP
#> 1 2012 DEU 3
#> 2 2010 AUT 4
#> 3 2011 AUT 5Zum Schluss kommen wir mit dplyr::nest_join() zu der komplexesten Funktion in der
*_join()-Familie.
Hier wird für jede Zeile im linken Datensatz in einer neuen Spalte ein ganzer
data.frame25 hinzugefügt,
der alle Zeilen vom rechten Datensatz enthält, die zu der entsprechenden linken Zeile passen:
data_bip_gini_nest_join <- dplyr::nest_join(
data_BIP, data_gini, by=c("Jahr"="year", "Land"="country"))
data_bip_gini_nest_join#> Jahr Land BIP data_gini
#> 1 2010 DEU 1 1
#> 2 2011 DEU 2 2
#> 3 2012 DEU 3
#> 4 2010 AUT 4
#> 5 2011 AUT 5
#> 6 2012 AUT 6 3In der Spalte y finden sich nun also sechs Data Frames (einer pro Zeile):
data_bip_gini_nest_join[["y"]] # die neue Spalte#> NULLJedes einzelne Element der Spalte y ist dabei ein eigener data.frame:
data_bip_gini_nest_join[["y"]][[1]] # erste Zeile der neuen Spalte#> NULLDas bedeutet, dass zur ersten Zeile des Datensatzes data_BIP aus dem Datensatz
data_gini genau eine Spalte passt und diese Spalte den Wert 1 enthält.
In der Praxis werden Sie nest_join() wenig verwenden, es ist wegen seiner
Flexibilität jedoch für das Programmieren extrem hilfreich.
Wie Sie vielleicht bemerkt haben, haben die Funktionen der *_join()-Familie
sehr ähnliche Argumente:
so verlangen alle *_join()-Funktionen als die ersten beiden Argumente
x und y zwei Datensätze, die als data.frame oder vergleichbares Objekt
vorliegen sollten, so wie data_BIP und data_Gini in unserem Beispiel.
Das dritte (optionale) Argument by, welches die ID-Spalten spezifiziert,
ist ebenfalls bei allen Funktionen gleich.
Achtung: wenn Sie by nicht explizit spezifizieren verwenden die Funktionen
alle Spalten mit gleichen Namen als ID-Spalten.
Zwar geben sie zur Info eine Warnung aus, aber Sie sollten das trotzdem
immer vermeiden und möglichst explizit sein.
Daher sollte by immer explizit gesetzt werden!
Ansonsten erhalten Sie (ohne Warnung) solche ungewünschten Ergebnisse:
debt_data_IWF <- data.frame(
Land=c("DEU", "GRC"), Schulden=c(10, 50))
debt_data_WELTBANK <- data.frame(
Land=c("GRC", "DEU"), Schulden=c(100, 25))
debt_data <- dplyr::full_join(debt_data_IWF, debt_data_WELTBANK)
debt_data#> Land Schulden
#> 1 DEU 10
#> 2 GRC 50
#> 3 GRC 100
#> 4 DEU 25Darüber hinaus findet sich das optionale Argument suffix sowohl bei
dplyr::inner_join(), dplyr::left_join(), dplyr::right_join() als auch dplyr::full_join().
Hier spezifizieren Sie eine Zeichenkette, die verwendet wird um Spalten, die in
beiden Datensätzen vorkommen, aber keine ID-Spalten sind, im gemeinsamen
Datensatz voneinander abzugrenzen. Standardmäßig ist dieses Argument auf
.x, .y eingestellt. Das bedeutet, dass wenn beide Datensätze eine Spalte Schulden
haben, diese aber nicht als ID-Spalte verwendet wird, beide Spalten als
Schulden.x und Schulden.y in den gemeinsamen Datensatz aufgenommen werden:
debt_data_IWF <- data.frame(
Land=c("DEU", "GRC"), Schulden=c(10, 50))
debt_data_WELTBANK <- data.frame(
Land=c("GRC", "DEU"), Schulden=c(100, 25))
debt_data <- dplyr::full_join(
debt_data_IWF, debt_data_WELTBANK, by=c("Land"))
debt_data#> Land Schulden.x Schulden.y
#> 1 DEU 10 25
#> 2 GRC 50 100Oder wir geben für den finalen Datensatz ein explizites suffix an, damit die
Variablennamen aussagekräftig werden:
debt_data <- dplyr::full_join(
debt_data_IWF, debt_data_WELTBANK, by=c("Land"),
suffix=c(".IWF", ".WELTBANK"))
debt_data#> Land Schulden.IWF Schulden.WELTBANK
#> 1 DEU 10 25
#> 2 GRC 50 100Tabelle 4.3 fasst die gerade diskutierten Funktionen noch einmal zusammen.
| Funktion | Effekt | Veränderung Anzahl Zeilen? |
|---|---|---|
left_join() |
x an y anhängen |
Unmöglich |
right_join() |
y an x anhängen |
Möglich |
inner_join() |
In x und y vorhandene Beobachtungen von y and x anhängen |
Reduktion möglich |
full_join() |
x und y kombinieren |
Vergrößerung möglich |
semi_join() |
Reduktion von x auf gemeinsame Beobachtungen |
Reduktion möglich |
anti_join() |
Reduktion von x auf ungeteilte Beobachtungen |
Reduktion möglich |
nest_join() |
Neue Spalte in x mit data.frame, der alle passenden Beobachtungen aus y enthält. |
Unmöglich |
Tipp: das Zusammenführen von Datensätzen ist extrem fehleranfällig. Häufig werden Probleme mit den Rohdaten hier offensichtlich. Daher ist es immer eine gute Idee, den zusammengeführten Datensatz genau zu inspizieren. Zumindest sollte man überprüfen ob die Anzahl an Zeilen so wie erwartet ist und ob durch das Zusammenführen Duplikate entstanden sind. Letzteres kann gerade in der Arbeit mit makroökonomischen Daten häufig vorkommen, wenn in einem Datensatz z.B. zwischen Ost-Deutschland und West-Deutschland unterschieden wird und man vorher die Namen aber in Länderkürzel überführt hat. In diesem Fall treten um 1990 herum häufig Duplikate auf. Damit kann man umgehen, man muss es aber erst einmal merken. Ich benutze z.B. immer die folgende selbst geschriebene Funktion um zu überprüfen ob es in einem neu generierten Datensatz Duplikate gibt:
#' Test uniqueness of data table
#'
#' Tests whether a data.table has unique rows.
#'
#' @param data_table A data frame or data table of which uniqueness should
#' be tested.
#' @param index_vars Vector of strings, which specify the columns of
#' data_table according to which uniqueness should be tested
#' (e.g. country and year).
#' @return TRUE if data_table is unique, FALSE and a warning if it is not.
#' @import data.table
test_uniqueness <- function(data_table, index_vars, print_pos=TRUE){
data_table <- data.table::as.data.table(data_table)
if (nrow(data_table)!=data.table::uniqueN(data_table, by = index_vars)){
warning(paste0("Rows in the data.table: ", nrow(data_table),
", rows in the unique data.table:",
data.table::uniqueN(data_table, by = index_vars)))
return(FALSE)
} else {
if (print_pos){
print(paste0("No duplicates in ", as.list(sys.call()[[2]])))
}
return(TRUE)
}
}Hier ein kleines Anwendungsbeispiel:
data_bip_gini_full_join <- dplyr::full_join(
data_BIP, data_gini, by=c("Jahr"="year", "Land"="country"))
test_uniqueness(
data_bip_gini_full_join, index_vars = c("Jahr", "Land"))#> [1] "No duplicates in data_bip_gini_full_join"#> [1] TRUEDie folgende Situation tritt häufiger auf: in den Daten werden für die Wendezeit getrennte Daten für West-Deutschland und das vereinigte Deutschland angegeben, aber die
countrycodeFunktion differenziert nicht zwischen den Namen wenn Sie sie in Ländercodes übersetzen. In der Folge entstehen Duplikate, die beim Zusammenführen der Daten dann offensichtlich werden (können):
bip_data#> Land Jahr BIP
#> 1 DEU 1989 1
#> 2 DEU 1990 2
#> 3 DEU 1991 3gini_data#> Land Jahr Gini
#> 1 West Germany 1989 1
#> 2 Germany 1989 2
#> 3 West Germany 1990 3
#> 4 Germany 1990 4
#> 5 Germany 1991 5gini_data <- dplyr::mutate(
gini_data, Land=countrycode(Land, "country.name", "iso3c"))
full_data <- dplyr::full_join(
bip_data, gini_data, by=c("Land", "Jahr"))
full_data#> Land Jahr BIP Gini
#> 1 DEU 1989 1 1
#> 2 DEU 1989 1 2
#> 3 DEU 1990 2 3
#> 4 DEU 1990 2 4
#> 5 DEU 1991 3 5test_uniqueness(full_data, index_vars = c("Land", "Jahr"))#> [1] FALSEAlternative in data.table: Eine Anleitung für das Zusammenführen von Datensätzem im
data.table-Format findet sich hier.
4.4.4 Datensätze filtern und selektieren
Sehr häufig haben Sie einen Rohdatensatz erhoben und benötigen für die weitere Analyse nur einen Teil dieses Datensatzes. Zwei Szenarien sind denkbar. Zum einen möchten Sie bestimmte Spalten nicht verwenden. Wir sprechen dann davon den Datensatz zu selektieren. Zum anderen möchten Sie bestimmte Zeilen nicht verwenden. Sie wollen nur Beobachtungen verwenden, die eine bestimmte Bedingung erfüllen, z.B. im Zeitraum 2012-2014 erhoben zu sein. In diesem Fall sprechen wir von filtern.
Wir lernen hier wie wir diese beiden Aufgaben mit den Funktionen filter() und
select() aus dem Paket dplyr, welches auch Teil des tidyverse ist, lösen
können.
Betrachten wir folgenden Beispieldatensatz:
data_al_exp_tidy#> # A tibble: 6 x 4
#> Land Jahr Exporte Arbeitslosigkeit
#> <chr> <chr> <dbl> <dbl>
#> 1 AT 2012 54.0 4.86
#> 2 AT 2013 53.4 5.34
#> 3 AT 2014 53.4 5.62
#> 4 DE 2012 46.0 5.38
#> 5 DE 2013 45.4 5.23
#> 6 DE 2014 45.6 4.98Um einzelne Spalten zu selektieren verwenden wir die Funktion dplyr::select().
Diese verlangt als erstes Argument den zu manipulierenden Datensatz und
danach die Namen oder Indices der Spalten, die behalten oder eliminiert
werden sollen. Spalten die behalten werden sollen werden einfach benannt,
bei Spalten, die eliminiert werden sollen schreiben Sie ein - vor den Namen:
head(
dplyr::select(data_al_exp_tidy, Land, Exporte),
2)#> # A tibble: 2 x 2
#> Land Exporte
#> <chr> <dbl>
#> 1 AT 54.0
#> 2 AT 53.4head(
dplyr::select(data_al_exp_tidy, -Exporte),
2)#> # A tibble: 2 x 3
#> Land Jahr Arbeitslosigkeit
#> <chr> <chr> <dbl>
#> 1 AT 2012 4.86
#> 2 AT 2013 5.34Häufig ist es besser die Namen der Spalten als character zu übergeben.
Das ist nicht nur besser lesbar, es wird später auch einfacher komplexere
Vorgänge zu programmieren indem Sie Funktionen schreiben, die den Namen von
Spalten als Argumente akzeptieren.
In diesem Fall können Sie wieder die Hilfsfunktion any_of() verwenden:
head(
dplyr::select(data_al_exp_tidy, any_of(c("Land", "Jahr"))),
2)#> # A tibble: 2 x 2
#> Land Jahr
#> <chr> <chr>
#> 1 AT 2012
#> 2 AT 2013head(
dplyr::select(data_al_exp_tidy, -any_of(c("Land", "Jahr"))),
2)#> # A tibble: 2 x 2
#> Exporte Arbeitslosigkeit
#> <dbl> <dbl>
#> 1 54.0 4.86
#> 2 53.4 5.34Tipp: Spalten auswählen: Die Funktion
any_of()erlaubt es Spalten mit sehr nützlichen Hilfsfunktionen auszuwählen. Manchmal möchten Sie z.B. alle Spalten auswählen, die mityear_anfangen, oder auf eine Zahl enden. Schauen Sie sich für solche Fälle einmal die select_helpers an, die auch weiter unten am Ende des Kapitels im Abschnitt 4.5 noch einmal aufgegriffen werden.
Wie im Abschnitt zu langen und weiten Daten bereits beschrieben
bietet sich in solchen Fällen die Pipe %>% an um Ihren Code zu vereinfachen und
besser lesbar zu machen. Es hat sich eingebürgert in die erste Zeile immer den
Ausgangsdatensatz zu schreiben und select dann in der nächsten Zeile mit implizitem
ersten Argument zu verwenden:
data_al_exp_selected <- data_al_exp_tidy %>%
dplyr::select(any_of(c("Land", "Jahr", "Exporte")))
head(data_al_exp_selected, 2)#> # A tibble: 2 x 3
#> Land Jahr Exporte
#> <chr> <chr> <dbl>
#> 1 AT 2012 54.0
#> 2 AT 2013 53.4Als nächstes wollen wir den Datensatz nach bestimmten Bedingungen filtern. Dabei ist es wichtig, die logischen Operatoren zu kennen, denn diese werden verwendet um Datensätze zu filtern.
Die Funktion dplyr::filter() akzeptiert als erstes Argument den Datensatz.
Wie oben folgen wir der Konvention das Argument in der Regel implizit über %>% zu
übergeben.
Danach können wir beliebig viele logische Abfragen, jeweils durch Komma getrennt,
an die Funktion übergeben.
Wenn wir z.B. nur Beobachtungen für Österreich nach 2012 im Datensatz belassen
wollen geht das mit:
data_al_exp_filtered <- data_al_exp_tidy %>%
dplyr::filter(
Land == "AT",
Jahr > 2012)
data_al_exp_filtered#> # A tibble: 2 x 4
#> Land Jahr Exporte Arbeitslosigkeit
#> <chr> <chr> <dbl> <dbl>
#> 1 AT 2013 53.4 5.34
#> 2 AT 2014 53.4 5.62Anstatt dem ,, welches implizit für & steht, können wir auch beliebig
komplizierte logische Abfragen einbauen. Wenn wir z.B. nur Beobachtungen wollen,
die für Österreich im Jahr 2012 oder 2014 und für Deutschland 2013 erhoben wurden
und in Deutschland zudem mit einer Arbeitslosigkeit über 5.3 % einhergehen,
geht das mit:
data_al_exp_filtered <- data_al_exp_tidy %>%
dplyr::filter(
(Land == "AT" & Jahr %in% c(2012, 2014)) | (Land=="DE" & Arbeitslosigkeit>5.3)
)
data_al_exp_filtered#> # A tibble: 3 x 4
#> Land Jahr Exporte Arbeitslosigkeit
#> <chr> <chr> <dbl> <dbl>
#> 1 AT 2012 54.0 4.86
#> 2 AT 2014 53.4 5.62
#> 3 DE 2012 46.0 5.38Zuletzt wollen wir noch sehen wie wir einzelne Spalten umbenennen können.
Das geht ganz einfach mit der Funktion dplyr::rename(), welche als erstes Argument
den Datensatz, und dann die Umbenennungsvorgänge in der Form Name_neu = Name_alt
verlangt.
Als Beispiel:
data_al_exp_tidy %>%
dplyr::rename(
country=Land,
year_observation=Jahr,
exports=Exporte,
unemployment=Arbeitslosigkeit)#> # A tibble: 6 x 4
#> country year_observation exports unemployment
#> <chr> <chr> <dbl> <dbl>
#> 1 AT 2012 54.0 4.86
#> 2 AT 2013 53.4 5.34
#> 3 AT 2014 53.4 5.62
#> 4 DE 2012 46.0 5.38
#> 5 DE 2013 45.4 5.23
#> 6 DE 2014 45.6 4.98Als abschließendes Beispiel kombinieren wir die neuen Funktionen und betrachten den Code, mit dem wir
aus dem Beispieldatensatz die Spalte zur Arbeitslosigkeit herausselektieren
nur die Beobachtungen für Deutschland nach 2012 betrachten und
die Spaltennamen dabei noch ins Englische übersetzen:
data_al_exp_tidy %>%
dplyr::select(
-any_of("Arbeitslosigkeit")
) %>%
dplyr::filter(
Jahr>2012,
Land=="DE"
) %>%
dplyr::rename(
country=Land,
year_observation=Jahr,
exports=Exporte)#> # A tibble: 2 x 3
#> country year_observation exports
#> <chr> <chr> <dbl>
#> 1 DE 2013 45.4
#> 2 DE 2014 45.6Alternative Implementierung mit
data.table: wie diese Operationen mit dem high-performance Paketdata.tabledurchgeführt werden können, wird hier sehr gut erläutert.
4.4.5 Datensätze zusammenfassen
In diesem letzten Abschnitt werden Sie lernen wie Sie Datensätze erweitern oder
zusammenfassen.
So können Sie eine neue Variable als eine Kombination bestehender
Variablen berechnen oder Ihren Datensatz zusammenfassen, z.B. indem Sie über alle
Beobachtungen über die Zeit für einzelne Länder den Mittelwert bilden. Zu diesem
Zweck werden wir hier die Funktionen mutate(), summarise() und group_by()
aus dem Paket dplyr
(Wickham u. a. 2019) verwenden.
Wir verwenden dplyr::mutate() um bestehende Spalten zu verändern oder neue Spalten zu
erstellen.
Betrachten wir dafür folgenden Beispieldatensatz:
head(unemp_data_wb)#> country year laborforce_female workforce_total population_total
#> 1: AT 2010 46.13933 4276558 8363404
#> 2: AT 2011 46.33455 4305310 8391643
#> 3: AT 2012 46.50653 4352701 8429991
#> 4: AT 2013 46.57752 4394285 8479823
#> 5: AT 2014 46.70688 4412800 8546356
#> 6: AT 2015 46.67447 4460833 8642699Angenommen wir möchten das Land mit den iso3c-Codes anstatt der iso2c-Codes
angeben, dann könnten wir mit der Funktion dplyr::mutate() die Spalte
country ganz einfach verändern:
unemp_data_wb2 <- unemp_data_wb %>%
dplyr::mutate(
country = countrycode::countrycode(country, "iso2c", "iso3c")
)
head(unemp_data_wb, 2)#> country year laborforce_female workforce_total population_total
#> 1: AT 2010 46.13933 4276558 8363404
#> 2: AT 2011 46.33455 4305310 8391643Links neben dem = steht der Name der neuen Spalte
(wenn der Name bereits existiert wird die existierende Spalte verändert).
Rechts neben dem = wird die Berechnung der neuen Werte spezifiziert.
Wie das Beispiel zeigt, kann hier durchaus der ursprüngliche Wert der Spalte
verwendet werden.
Wir können mit dplyr::mutate() aber auch einfach neue Spalten erstellen, wenn der
Name links vom = noch nicht als Spalte im Datensatz existiert.
Wenn Sie nun z.B. wissen möchten, wie viele Frauen absolut in Deutschland und
Österreich zur Erwerbsbevölkerung gehören, müssen wir den prozentualen Anteil mit
der Anzahl an Erwerbstätigen multiplizieren.
Das bedeutet, wir müssen die Spalten laborforce_female und workforce_total
multiplizieren und durch 100 Teilen, da laborforce_female in Prozent angegeben ist.
Das machen wir mit der Funktion dplyr::mutate(), wobei wir eine neue
Spalte mit dem Namen workers_female_total erstellen wollen:
unemp_data_wb <- unemp_data_wb %>%
dplyr::mutate(
workers_female_total = laborforce_female*workforce_total/100
)
head(unemp_data_wb, 2)#> country year laborforce_female workforce_total population_total
#> 1: AT 2010 46.13933 4276558 8363404
#> 2: AT 2011 46.33455 4305310 8391643
#> workers_female_total
#> 1: 1973175
#> 2: 1994846Vielleicht sind wir für unseren Anwendungsfall gar nicht so sehr an der
Veränderung über die Zeit interessiert, sondern wollen die
durchschnittliche Anzahl an Frauen in der Erwerbsbevölkerung berechnen.
Das würde bedeuten, dass wir die Anzahl der Spalten in unserem Datensatz
reduzieren - etwas das bei der Anwendung von dplyr::mutate() nie passieren
würde.
Dafür gibt es die Funktion dplyr::summarise():26
unemp_data_wb_summarized <- unemp_data_wb %>%
dplyr::summarise(
fem_workers_avg = mean(workers_female_total)
)
unemp_data_wb_summarized#> fem_workers_avg
#> 1 10761223Wie Sie sehen, funktioniert die Syntax quasi äquivalent zu dplyr::mutate(), allerdings
kondensiert dplyr::summarise() den gesamten Datensatz auf die definierte Zahl.
Im gerade berechneten Durchschnitt sind sowohl die Werte für Deutschland als auch
Österreich eingegangen. Das erscheint erst einmal irreführend, es wäre wohl
besser einen Durchschnittswert jeweils für Deutschland und Österreich getrennt
zu berechnen.
Das können wir erreichen, indem wir den Datensatz vor der Anwendung von
dplyr::summarise() gruppieren.
Das funktioniert mit der Funktion dplyr::group_by(), die als Argumente die
Spalten, nach denen wir gruppieren wollen, akzeptiert.
Sie sollten sich in jedem Fall angewöhnen, nach dem Gruppieren den Datensatz mit
dplyr::ungroup() wieder in den ursprünglichen Zustand zurückzuführen:
unemp_data_wb %>%
dplyr::group_by(country) %>%
dplyr::summarise(
fem_workers_avg = mean(workers_female_total)
) %>%
dplyr::ungroup()#> # A tibble: 2 x 2
#> country fem_workers_avg
#> <chr> <dbl>
#> 1 AT 2042685.
#> 2 DE 19479761.Natürlich können Sie dplyr::group_by() auch im Zusammenhang mit dplyr::mutate() oder anderen
Funktionen verwenden.
Wie Sie sehen ist der Effekt aber durchaus unterschiedlich:
unemp_data_wb %>%
dplyr::group_by(country) %>%
dplyr::mutate(
fem_workers_avg = mean(workers_female_total)
) %>%
dplyr::ungroup()#> # A tibble: 14 x 7
#> country year laborforce_fema… workforce_total population_total
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 AT 2010 46.1 4276558 8363404
#> 2 AT 2011 46.3 4305310 8391643
#> 3 AT 2012 46.5 4352701 8429991
#> 4 AT 2013 46.6 4394285 8479823
#> 5 AT 2014 46.7 4412800 8546356
#> 6 AT 2015 46.7 4460833 8642699
#> 7 AT 2016 46.7 4531193 8736668
#> 8 DE 2010 45.6 42014274 81776930
#> 9 DE 2011 45.9 41674901 80274983
#> 10 DE 2012 45.9 41767969 80425823
#> 11 DE 2013 46.1 42161170 80645605
#> 12 DE 2014 46.2 42415215 80982500
#> 13 DE 2015 46.3 42731868 81686611
#> 14 DE 2016 46.4 43182140 82348669
#> # … with 2 more variables: workers_female_total <dbl>, fem_workers_avg <dbl>Der Datensatz wird nicht verkleinert und keine Spalte geht verloren.
Dafür wiederholen sich die Werte in der neu geschaffenen Spalte.
Je nach Anwendungsfall ist also die Verwendung von dplyr::mutate() oder
dplyr::summarise() im Zusammenspiel mit dplyr::group_by() angemessen.
Im Folgenden werden wir uns noch ein etwas komplexeres Beispiel anschauen:
wir werden zunächst die jährliche Veränderung in der absoluten Anzahl der
weiblichen Erwertbstätigen in Österreich und Deutschland ermitteln und
dann vergleichen ob dieser Wert größer ist als das Bevölkerungswachstum in dieser
Zeit.
Dazu verwenden wir die Funktion dplyr::lag() um den Wert vor dem aktuellen
Wert zu bekommen.27
Zuletzt wollen wir nur noch die berechneten Spalten im Datensatz behalten.
unemp_data_wb_growth <- unemp_data_wb %>%
dplyr::group_by(country) %>%
dplyr::mutate(
pop_growth=(
population_total-lag(population_total))/lag(population_total),
fem_force_growth=(
workers_female_total-lag(workers_female_total))/lag(workers_female_total)
) %>%
dplyr::ungroup() %>%
dplyr::mutate(
fem_force_growth_bigger=fem_force_growth>pop_growth
) %>%
dplyr::select(
any_of(c("country", "year", "pop_growth",
"fem_force_growth", "fem_force_growth_bigger"))
)
unemp_data_wb_growth#> # A tibble: 14 x 5
#> country year pop_growth fem_force_growth fem_force_growth_bigger
#> <chr> <dbl> <dbl> <dbl> <lgl>
#> 1 AT 2010 NA NA NA
#> 2 AT 2011 0.00338 0.0110 TRUE
#> 3 AT 2012 0.00457 0.0148 TRUE
#> 4 AT 2013 0.00591 0.0111 TRUE
#> 5 AT 2014 0.00785 0.00700 FALSE
#> 6 AT 2015 0.0113 0.0102 FALSE
#> 7 AT 2016 0.0109 0.0166 TRUE
#> 8 DE 2010 NA NA NA
#> 9 DE 2011 -0.0184 -0.00206 TRUE
#> 10 DE 2012 0.00188 0.00311 TRUE
#> 11 DE 2013 0.00273 0.0145 TRUE
#> 12 DE 2014 0.00418 0.00813 TRUE
#> 13 DE 2015 0.00869 0.00993 TRUE
#> 14 DE 2016 0.00810 0.0126 TRUE4.5 Gleichzeigite Bearbeitung mehrerer Spalten
Häufig müssen wir mehrere Spalten mit den gleichen Funktionen bearbeiten.
In diesem Kontext sind die so genannten
select_helpers
in Kombination mit der Funktion dplyr::across(), die mit Version 1.0 in
das Paket dplyr eingeführt wurde, sehr
hilfreich.
Hier möchte ich diese Funktionen anhand eines kurzen Beispiels einführen,
verweise aber für eine detailliertere Beschreibung auf die entsprechenden
offiziellen Vignetten (siehe
hier und
hier.
)
Betrachten wie also folgendes Beispiel:
#> iso3c year group AIS_mean AIS_MIP AIS_SGP AIS_INT SGP_new
#> 1: FRA 2012 Periphery 0.50 0.5833333 0.5 0.2500000 0.1738386
#> 2: SVN 2012 Catchup 0.50 0.5000000 0.5 0.5000000 0.1627661
#> 3: FIN 2012 Core 0.55 0.5000000 0.5 0.5833333 0.4861746
#> SGP_avg
#> 1: 0.7324741
#> 2: 0.4489916
#> 3: 0.1890547Dieser Datensatz enthält Informationen wie Länder (iso3c) bestimmte Vorgaben
der EU implementieren. Die Spalte group gibt die Gruppe an, zu der das Land
gehört, die weiteren Spalten enthalten Informationen über die allgemeine
Implementierung von Vorgaben (‘AIS’ steht für ‘Average Implementation Score’)
aus bestimmten Bereichen.
Der Inhalt ist jedoch nicht zentral, vielmehr wollen wir anhand des Datensatzes
bestimmte Aufbereitungsstrategien illustrieren.
Die select_helpers erlauben es uns bestimmte Spalten auszuwählen.
Wir haben einen dieser Helper bereits kennen gelernt - dplyr::any_of().
Insgesamt gibt es 9 diesen Hilfsfunktionen, die in Tabelle 4.4
zusammengefasst sind.
| Funktion | Funktion |
|---|---|
everything() |
Alle Spalten |
last_col() |
Wählt die letzte Spalte |
starts_with() |
Alle Spalten, die mit einem bestimmten Prefix starten |
ends_with() |
Alle Spalten, die mit einem bestimmten Sufffix enden |
contains() |
Alle Spalten, die einen bestimmten String enthalten |
matches() |
Sucht mit Hilfe regulärer Ausdrücke (‘regular expressions’) |
num_range() |
Spalten, die einem numerischen Muster folden |
all_of() |
Spalten, die in einem Vektor genannt sind |
any_of() |
Wie all_of(), aber ohne Fehler wenn nicht alle Spalten vorkommen |
Das Prinzip ist immer gleich, hier wollen wir ihre Funktion anhand von
dplyr::starts_with() illustrieren. Nehmen wir an, wir wollen alle Spalten auswählen,
die mit AIS beginnen. In diesem Fall verwenden wir die Funktion
dplyr::starts_with() in Kombination mit dplyr::select():
ais_subset1 <- eu_ais_data %>%
dplyr::select(dplyr::starts_with("AIS"))
head(ais_subset1, 3)#> AIS_mean AIS_MIP AIS_SGP AIS_INT
#> 1: 0.50 0.5833333 0.5 0.2500000
#> 2: 0.50 0.5000000 0.5 0.5000000
#> 3: 0.55 0.5000000 0.5 0.5833333Eventuell wollen wir auch die Informationen über das Beobachtungsjahr und das
Land behalten. Dann können wir die select_helpers auch kombinieren:
ais_subset1 <- eu_ais_data %>%
dplyr::select(
dplyr::any_of(c("iso3c", "year")),
dplyr::starts_with("AIS")
)
head(ais_subset1, 3)#> iso3c year AIS_mean AIS_MIP AIS_SGP AIS_INT
#> 1: FRA 2012 0.50 0.5833333 0.5 0.2500000
#> 2: SVN 2012 0.50 0.5000000 0.5 0.5000000
#> 3: FIN 2012 0.55 0.5000000 0.5 0.5833333Mit den select_helpers aus Tabelle 4.4 können Sie eigentlich
jede beliebige Menge an Spalten auswählen.
Das wird besonders hilfreich wenn wir die Auswahl von Spalten über die Funktion
dplyr::across() mit anderen Funktionen aus dem tidyverse kombinieren.
Nehmen wir z.B. an wir wollen den Mittelwert von allen Spalten bilden, die
mit ‘AIS’ anfangen und dabei die Daten pro Jahr gruppieren:
eu_ais_data %>%
dplyr::group_by(year) %>%
dplyr::summarise(
dplyr::across(
dplyr::starts_with("AIS"), ~mean(., na.rm = TRUE)),
.groups="drop")#> # A tibble: 5 x 5
#> year AIS_mean AIS_MIP AIS_SGP AIS_INT
#> <int> <dbl> <dbl> <dbl> <dbl>
#> 1 2012 0.555 0.493 0.667 0.535
#> 2 2013 0.424 0.292 0.667 0.473
#> 3 2014 0.411 0.383 0.375 0.451
#> 4 2015 0.455 0.5 0.5 0.5
#> 5 2016 0.339 0.375 0.25 0.25Das erste Argument zu dplyr::across() ist der Selektor.
Die Funktion kann auf verschiedene Arten übergeben werden, hier habe ich mich
für die Notation mit ~ entschieden, die es einfach macht, noch zusätzliche
Argumente für die Funktion zu spezifizieren.
Das finale Argument .groups gehört zur Funktion dplyr::summarise() und
macht das Auflösen der Gruppierung explizit, ist aber nicht absolut notwendig.
Wir können auch mehrere Funktionen auf einmal verwenden und die Namen der neuen Spalten manuell festlegen. Das ist hilfreich wenn wir z.B. nicht nur die Mittelwerte, sonder auch die Standardabweichung berechnen wollen. Dazu bietet sich folgende Schreibweise an:
eu_ais_data %>%
dplyr::group_by(group) %>%
summarise(
across(contains("SGP"),
list(mean = mean, sd = sd),
.names = "{.col}-{.fn}"),
.groups="drop"
)#> # A tibble: 4 x 7
#> group `AIS_SGP-mean` `AIS_SGP-sd` `SGP_new-mean` `SGP_new-sd` `SGP_avg-mean`
#> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 Catc… 0.438 0.315 0.309 0.360 0.451
#> 2 Core 0.625 0.212 0.521 0.252 0.574
#> 3 Fina… 0.458 0.246 0.576 0.253 0.450
#> 4 Peri… 0.5 NA 0.174 NA 0.732
#> # … with 1 more variable: `SGP_avg-sd` <dbl>Hier haben wir mit dem Argumetn .names auch die Namen der neuen Spalten
festgelegt. Wie das Beispiel zeigt ist die Funktion hier extrem flexibel und
erlaubt die Verwendung der so genannten glue-Schreibweise - Details
finden Sie bei Bedarf hier.
Hilfreich ist häufig auch die Kombination mit der Funktion dplyr::where(),
die es uns erlaubt eine Funktion auf alle Spalten anzuwenden, für die ein
Test den Wert TRUE ausgibt.
Das ist z.B. hilfreich, wenn wir eine Funktion auf alle Spalten anwenden
wollen, die Input eines bestimmten Typs (z.B. character) enthalten.
Der folgende Code zählt die Anzahl der eindeutigen Werte für alle Spalten
vom Typ character:
eu_ais_data %>%
summarise(across(where(is.character), ~length(unique(.))))#> iso3c group
#> 1 10 4Und der folgende Code berechnet den Mittelwert für alle Spalten vom Typ
double:
eu_ais_data %>%
summarise(across(where(is.double), ~mean(., na.rm=T)))#> AIS_mean AIS_MIP AIS_SGP AIS_INT SGP_new SGP_avg
#> 1 0.4511416 0.3933485 0.5357143 0.4752976 0.4797278 0.5226087Falls Sie übrigens die Mittelwerte über Spalten hinweg berechnen wollen, also
eine Operation zeilenweise durchführen wollen, dann können Sie die die Variante
dplyr::c_across() mit der Funktion dplyr::rowwise() kombinieren:
eu_ais_data %>%
rowwise() %>%
dplyr::mutate(
ais_mean = mean(c_across(dplyr::starts_with("AIS")))
) %>%
head(3)#> # A tibble: 3 x 10
#> # Rowwise:
#> iso3c year group AIS_mean AIS_MIP AIS_SGP AIS_INT SGP_new SGP_avg ais_mean
#> <chr> <int> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 FRA 2012 Periphe… 0.5 0.583 0.5 0.25 0.174 0.732 0.458
#> 2 SVN 2012 Catchup 0.5 0.5 0.5 0.5 0.163 0.449 0.5
#> 3 FIN 2012 Core 0.55 0.5 0.5 0.583 0.486 0.189 0.533Grundsätzlich sind Berechnungen über Spalten hinweg immer umständlicher und
häufig sollte man sich überlegen nicht einen Umweg über tidyr::pivot_longer()
and tidyr::pivor_wider() zu gehen und die Operationen spaltenweise auszuführen.
4.6 Abschließende Bemerkungen zum Umgang mit Daten innerhalb eines Forschungsprojekts
Das zentrale Leitmotiv dieses Kapitels war die Idee, dass die Datenaufbereitung vom ersten Schritt an reproduzierbar und transparent sein sollte. Wenn Sie gefragt werden, wie Ihre Ergebnisse zustande gekommen sind, sollten Sie in der Lage sein, jeden einzelnen Arbeitsschritt seit der ersten Akquise der Daten offenzulegen, bzw. nachvollziehbar zu machen.
Es ist ein zentraler Nachteil von point-and-click-Software (wie z.B. SPSS),
dass Sie für eine Reproduktion jeden einzelnen Mausklick vor dem Rechner
wiederholen, bzw. erklären müssten.
Zum Glück ist das mit Skript-basierten Sprachen wie R anders:
Sie können einfach ein Skript Datenaufbereitung.R anlegen, in welchem Sie die aus
dem Internet heruntergeladenen Daten in den für die Analyse aufbereiteten Datensatz
umwandeln.
Wenn jemand wissen möchte, wo die Daten herkommen, die Sie in Ihrer Analyse verwenden,
brauchen Sie der Person nur die Quelle der Daten zu nennen und ihr
Skript zu zeigen.
So ist es für Sie auch leicht Ihre Analyse mit neuen Daten zu aktualisieren.
Daher hat sich in der Praxis häufig die in Abbildung 4.5 aufgezeigte oder eine ähnliche Ordnerstruktur bewährt:

Abbildung 4.5: Bewährte Ordnerstruktur für Forschungsprojekte mit R.
Der Vorteil an dieser Ordnerstruktur ist, dass Sie die Rohdaten in einem separaten Ordner gespeichert haben und so explizit vom Rest Ihres Workflows abgrenzen. Denn: Rohdaten sollten nie bearbeitet werden. Zu leicht gerät in Vergessenheit welche Änderungen tatsächlich vorgenommen wurden und Ihre Forschung wird dadurch nicht mehr replizierbar - weder für Sie noch für andere. Alle weiteren Änderungen an den Rohdaten sollten über ein Skript vorgenommen werden, sodass immer klar ist wie Sie von den Rohdaten zu den Analysedaten kommen.
Diese bearbeiteten Daten können in einem zweiten Unterordner (hier: tidy)
gespeichert werden, damit Sie für Ihre Analyse nicht immer die Daten neu
aufbereiten müssen.
Gerade bei großen Datensätzen kann das nämlich sehr lange dauern.
Wichtig ist aber, dass die Daten in tidy immer mit Hilfe eines Skripts aus den
Daten in raw wiederhergestellt werden können.
In der Praxis würden Sie also aus den Daten in raw, die entweder direkt aus dem
Internet geladen wurden oder direkt aus einem Experiment hervorgegangen sind, per
Skript Datenaufbereitung.R den Datensatz AufbereiteteDaten.csv erstellen.
Dabei können auch mehrere Rohdatensätze zusammengeführt werden.
Dieser Datensatz kann dann in der weiteren Analyse verwendet werden, z.B. im Skript
StatistischeAnalyse.R, das dann einen Output in Form einer Datei
WunderbarerErgebnisplot.pdf produziert.
Der Vorteil: wenn jemand genau wissen möchte, wie WunderbarerErgebnisplot.pdf
produziert wurde können Sie sämtliche Schritte ausgehend von den vollkommen
unangetasteten Rohdaten transparent machen.
Durch die Trennung unterschiedlicher Arbeitsschritte - wie Datenaufbereitung und
statistische Analyse - bleibt Ihr Projekt zudem übersichtlich.
4.7 Anmerkungen zu Paketen
In diesem Kapitel wurden gleich mehrere Pakete aus dem
tidyverse, einer Sammlung von Paketen, verwendet.
Zwar schätze ich das tidyverse sehr, gleichzeitig ist der Fokus von R Studio auf
diese Pakete zumindest potenziell problematisch.
Dies wird in diesem kritischen Blogpost
sehr schön beschrieben.
Was die Einsteigerfreundlichkeit vom tidyverse angeht, bin ich jedoch anderer
Meinung als der Verfasser des Blogposts: meiner Meinung nach machen diese Pakete die Arbeit mit
Datensätzen sehr einfach, und für kleine Datensätze (<500MB) benutze ich das
tidyverse auch in meiner eigenen Forschung.
Es sollte jedoch klar sein, dass es nur eine Option
unter mehreren ist, weswegen ich versuche in meinen Paketen vollständig auf
das tidyverse zu verzichten - auch weil es in puncto Performance deutlich
schlechter ist als z.B. data.table
(Dowle und Srinivasan 2019), das auch für mehrere hundert GB große Datensätze gut geeignet ist.
Zur Aneignung des Pakets
data.table (Dowle und Srinivasan 2019)
ist das offizielle Tutorial
gut geeignet, macht m.E. aber auch deutlich,
dass es für die ersten Schritte mit R etwas unintuitiver ist als das tidyverse.
Wenn Sie später einmal beide Ansätze beherrschen, können Sie das tun, was in einer diversen Sprache wie R das einzig Richtige ist: je nach Anwendungsfall das passende Paket wählen - ganz wie im Falle von Paradigmen in einer Pluralen Ökonomik.
Das klassische Beispiel in der Psychologie ist ‘Intelligenz’.↩︎
Interessanterweise hat der maßgeblich an der Entwicklung des Indikators BIP beteiligte Simon Kuznets in Kuznets (1934) davon abgeraten, diese Operationalisierung als Indikator für wirtschaftliche Entwicklung zu verwenden. Für mehr Infos dazu siehe z.B. Lepenies (2016).↩︎
Allerdings müssen wir bei der Funktion noch das Argument
type=1odertype=3ergänzen, um einen Quantilsalgorithmus zu wählen, der auch mit Faktoren funktioniert. Eine Beschreibung der unterschiedlichen Algorithmen finden Sie überhelp(quantile).↩︎Das bedeutet natürlich weder, dass Sie (a) diesen Daten blind vertrauen sollten, noch dass (b) Ihre Daten tatsächlich die latente Variable messen, an der Sie interessiert sind. Häufig besteht großer Dissens mit welchem Maß welche latente Variable gemessen werden kann. Entsprechend geht der Auswahl der Daten häufig viel Zeit des theoretischen Überlegens voraus. Hier gehen wir davon aus, dass Sie sich über die richtigen Daten schon im Klaren sind.↩︎
Da ein solcher Code nur funktioniert wenn Sie mit dem Internet verbunden sind und Sie die Daten ja nicht jedes Mal von neuem herunterladen wollen macht es Sinn, die Daten nach dem Runterladen abzuspeichern, auch um den konkreten Datensatz, mit dem Sie Ihre Ergebnisse bekommen haben, zu konservieren.↩︎
Zwar gibt es im
WDI-Paket auch die FunktionWDI::WDIsearch(), mit der Sie Datensätze direkt suchen können, allerdings funktioniert das meiner Erfahrung nach nicht optimal.↩︎Auch hier gilt, dass die automatische Erkennung von
data.table::fread()schon sehr gut funktioniert, aber die manuelle Eingabe immer sicherer und transparenter ist.↩︎Wenn Sie das Paket
bit64nicht installiert haben, bekommen Sie zudem eine etwas merkwürdig anmutende Warnung zu lesen.↩︎Wie hier beschrieben ist das Konzept von ‘tidy data’ nicht neu: Statistiker*innen sprechen bei einem ‘tidy’ Datensatz häufig von einer ‘Datenmatrix’. Wer sich mehr mit der zugrundeliegenden Theorie beschäftigen möchte sollte zunächst die 12 Regeln von Edgar Codd und ihre Begründung nachlesen.↩︎
Das steht nicht im Widerspruch zu dem oben formulierten Ziel am Ende
tidyDaten zu haben. Es ist nur so, dass Sie für manche statistischen Transformationen als Zwischenschritt in ein anderes Format wechseln müssen, oder der Weg hin zutidyDaten das Wechseln zwischen langen und breiten Datensätzen erforderlich macht. Das wird durch die Beispiele später in diesem Kapitel praktisch deutlich werden.↩︎Die Funktionen
pivot_longer()undpivot_wider()wurden in der neuesten Version vontidyreingeführt. Achten Sie also darauf, dass Sie die neueste Version installiert haben. Sie ersetzen die Funktionentidyr::spread()undtidyr::gather(), die natürlich noch weiterhin funktionieren und die Sie in älterem Code sicher noch häufig finden werden. In diesem Blog-Post beschreibt Chefentwickler Hadley Wickham die neuen Funktionen und grenzt sie von den älteren Implementierungen ab.↩︎Die Funktionen
dplyr::summarize()unddplyr::summarise()sind Synonyme.↩︎Es gibt neben den Funktionen
dplyr::lag()unddplyr::lead()auch die Funktionendplyr::first()unddplyr::last(), die Sie verwenden können um Änderungen über den gesamten Zeitraum zu berechnen. Achten Sie jedoch auf den möglichen Konflikt zwischen den Funktionendata.table::first()unddplyr::first()sowiedata.table::last()unddplyr::last()!↩︎